Metagenome Sequencing
採用新一代高通量NGS次世代定序技術,以特定環境中的整個微生物群落作為研究對象,獲得環境微生物基因資訊總和,研究微生物的群落結構、物種分類、系統進化、基因功能及代謝網路等。
總體基因體定序擺脫了傳統研究中微生物分離培養的技術限制,直接萃取環境樣品DNA進行定序,有通量高、速度快、訊息全面等特點,在鑑定低豐富度的微生物群落及挖掘更多基因資源方面具有很大的優勢,基於定序技術和生物資訊學的快速發展,總體基因體技術優勢在微生物研究領域中愈來愈明顯,目前已廣泛應用於各種環境體系微生物研究中。
全面性微生物功能挖掘
• 物種、基因注釋
• 基因預測
• 功能富集統計、聚類分析
• 代謝網路
加入詢價車
總體基因體定序擺脫了傳統研究中微生物分離培養的技術限制,直接萃取環境樣品DNA進行定序,有通量高、速度快、訊息全面等特點,在鑑定低豐富度的微生物群落及挖掘更多基因資源方面具有很大的優勢,基於定序技術和生物資訊學的快速發展,總體基因體技術優勢在微生物研究領域中愈來愈明顯,目前已廣泛應用於各種環境體系微生物研究中。
全面性微生物功能挖掘
• 物種、基因注釋
• 基因預測
• 功能富集統計、聚類分析
• 代謝網路
分析流程
基於Illumina NovaSeq 6000定序平台,不經由PCR放大或篩選將DNA樣品建庫定序。總體基因體定序資料可進行微生物群體的組成與α、β多樣性、交互作用、代謝路徑變化、基因功能差異等分析。
完整分析流程包含:品質控管、組裝、序列分群(Binning/CAGs)、基因預測、物種注釋、多變量統計、差異統計、功能資料庫分析(COG (蛋白質功能)、eggNOG (蛋白質功能)、KEGG (基因功能與代謝途徑)、CARD (細菌抗藥性基因)、VFDB (毒力因子)、NCyc (氮循環)、dbCAN2 (碳水化合物酵素)等) 。
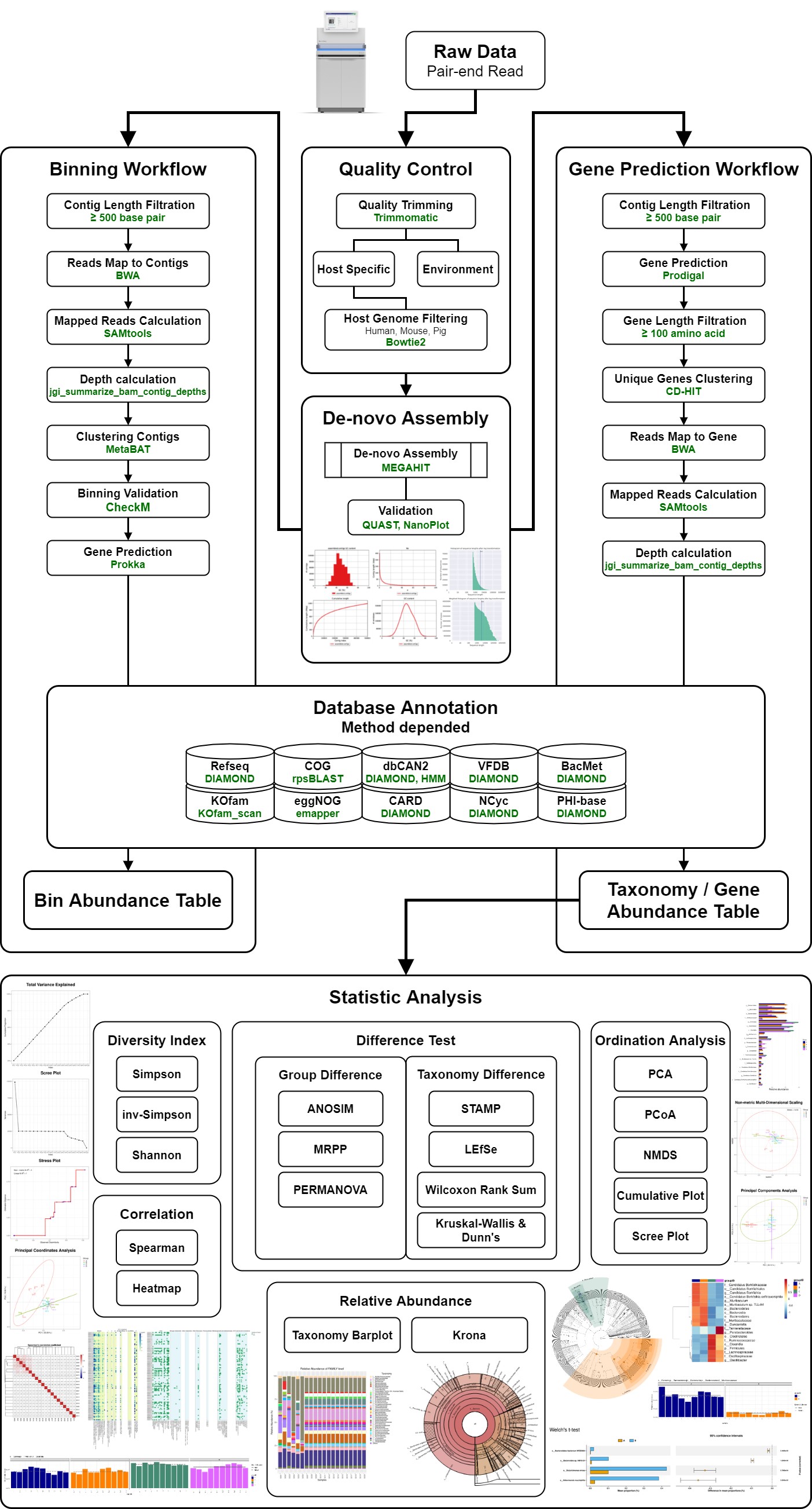
完整分析流程包含:品質控管、組裝、序列分群(Binning/CAGs)、基因預測、物種注釋、多變量統計、差異統計、功能資料庫分析(COG (蛋白質功能)、eggNOG (蛋白質功能)、KEGG (基因功能與代謝途徑)、CARD (細菌抗藥性基因)、VFDB (毒力因子)、NCyc (氮循環)、dbCAN2 (碳水化合物酵素)等) 。

服務流程
樣品通過檢測後,40個工作天(含分析)
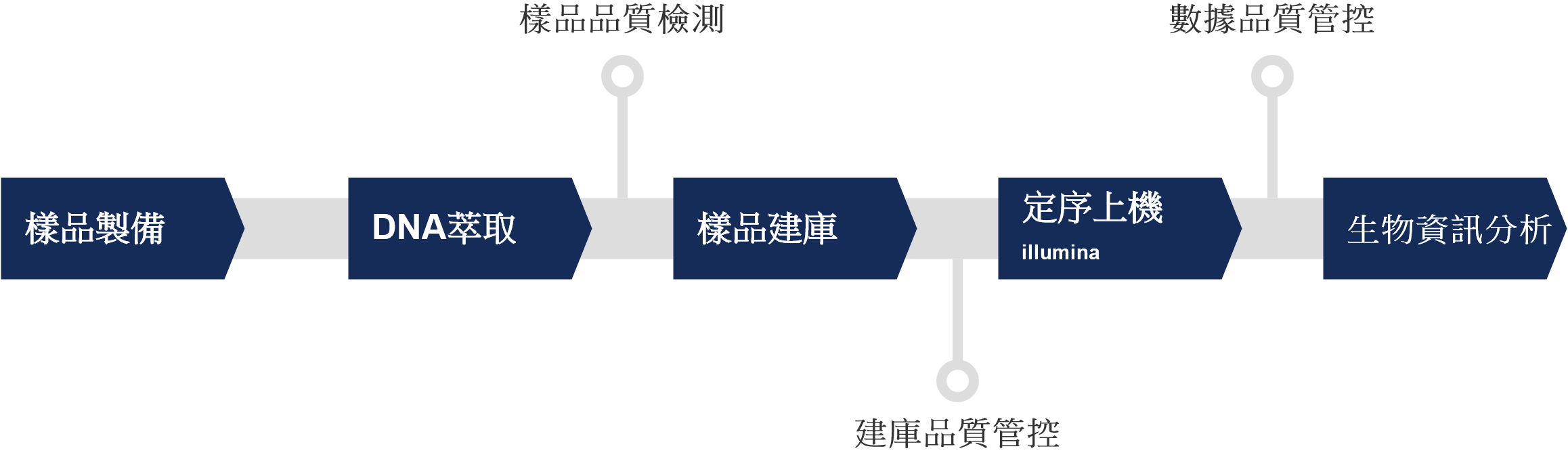

樣品需求
- 樣品總量:
DNA 總量: ≧ 1 ug (Qubit®測定)
DNA 濃度: ≧ 50 ng/ ul (Qubit®測定)
樣品體積: ≧ 20 ul
※樣品濃度定量Qubit®測定為主
- 樣品純度:
OD260/OD280 = 1.8-2.0
DNA無降解且無汙染
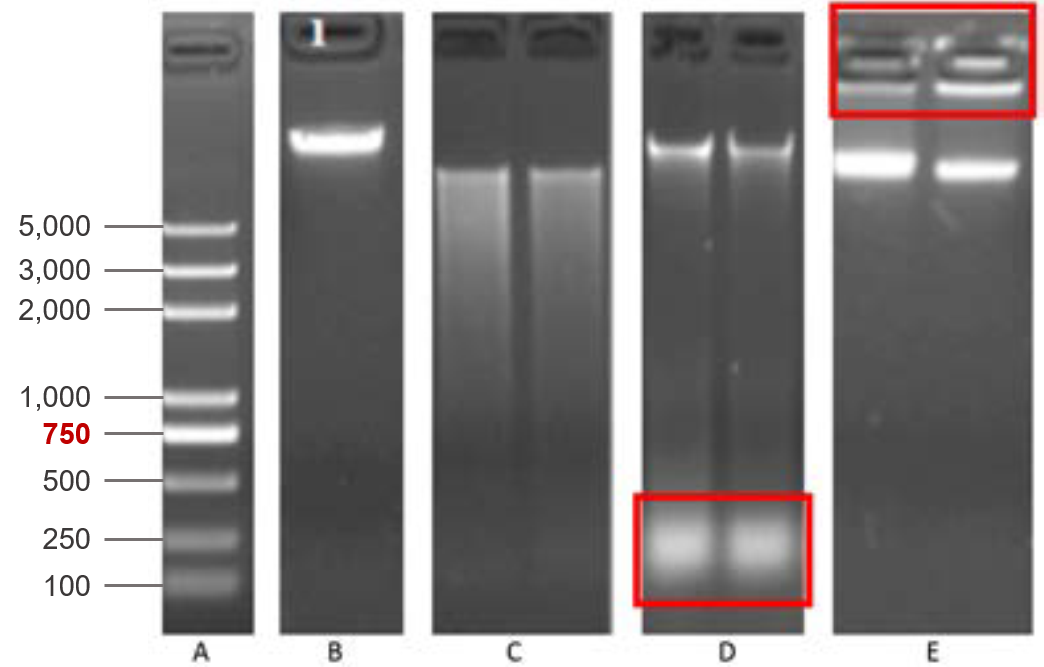
建議先行以電泳膠圖確認片段大小及DNA完整度,並於送件時提供膠圖結果
(A) DNA Marker, (B) 合格樣品 (C) 降解樣品
(D) RNA汙染樣品 (E) 蛋白質殘留汙染樣品 (以紅框標記汙染處)
定序規格
Novaseq 6000, paired-end 150 bp
採樣建議
糞便
1.<建議>新鮮檢體,1 g (建議3-5 g),-80度凍存
2.保存液,1 mL (建議3-5 mL),直接將檢體與保存液均質,可常溫或凍存運送
口腔樣品
Swab - 無保存液,刮取口腔黏膜後以無菌剪刀將採樣端剪下冷凍保存於eppendorf,-80度凍存
皮膚、生殖道
Swab - 無保存液,刮取欲採檢區域後以無菌剪刀將採樣端剪下冷凍保存於eppendorf,-80度凍存
底泥、土壤
新鮮檢體,採樣後進行混勻1 g (建議3-5 g)置入eppendorf,-80度凍存
水體樣品
離心pellet,-80度凍存 (由於環境水樣品較為多元,採集方式也有差異,請先聯繫業務進行後續討論)
其他注意事項
1.若為腸道內容物採樣,需將腸道分離,內容物取出冷凍保存
2.採樣棉棒建議以無菌剪刀將採樣端剪下後保存
3.環境樣品建議在採樣後先進行混勻後分裝於1.5 mL微量離心管