10x Genomics Single Cell Gene Expression Flex
搭配10x Genomics Chromium X 平台將細胞簡易處理即可上機定序,擴展技術應用的範圍,為單細胞RNA定序技術的未來創造更多的可能性。(目前僅適用於人類及小鼠)
技術原理
Probe可以專一性辨認基因序列,成對的Probe由LHS/ RHS組成,Probe barcode最高有16種組合,可以用來區分樣品。 (10x Genomics)
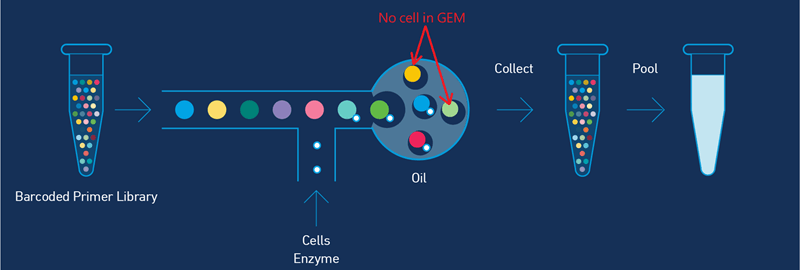
使用Chromium Single Cell Gene Expression,結合10x Genomics 獨家專利GemCode™油滴微珠技術區分單一細胞,並將帶有特定Barcode序列的微珠一同全數包入Droplet內,完成反轉錄即可接續擴增建庫流程,搭配使用NovaSeq X, paired-end 150 bp
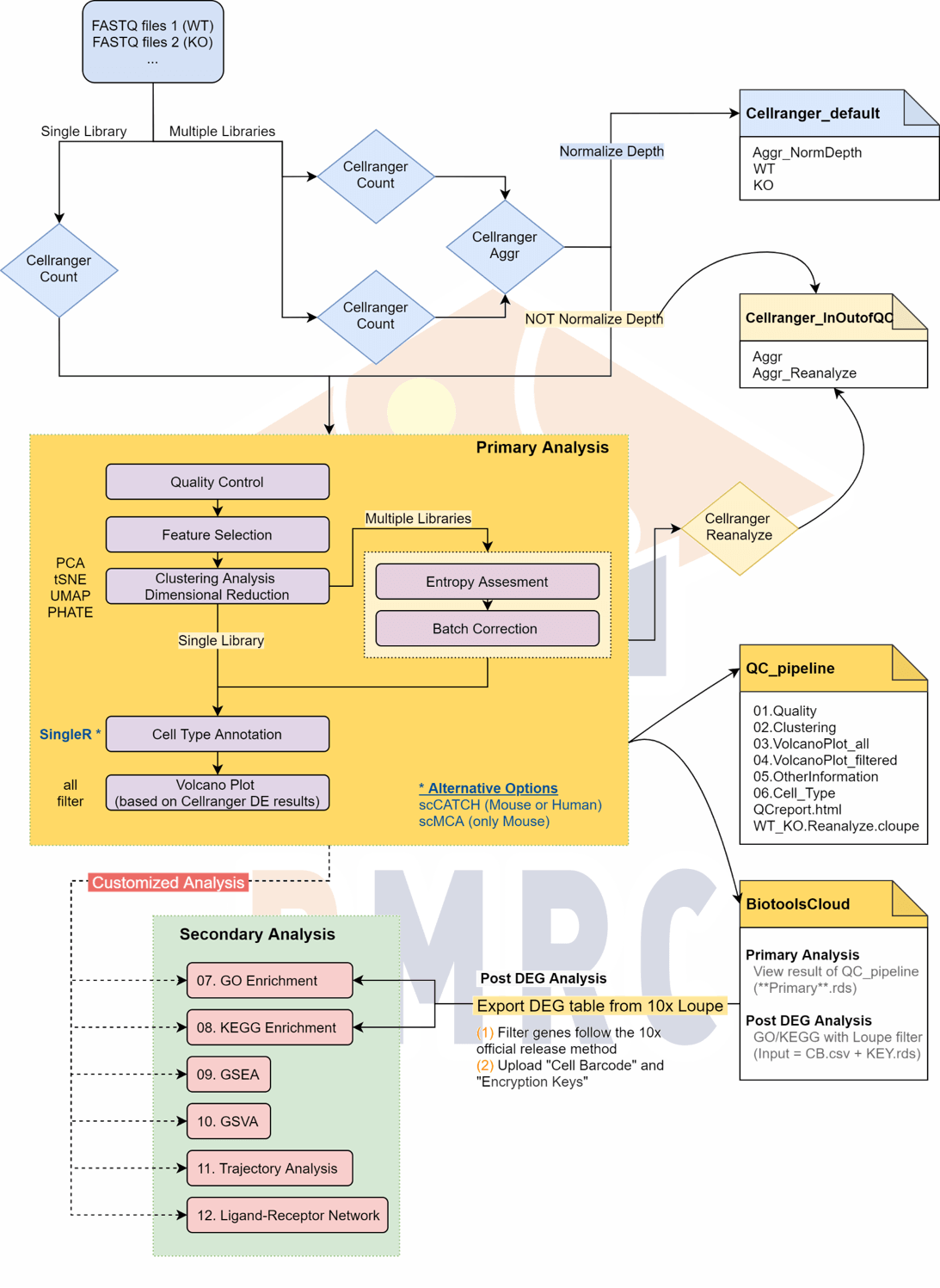
Single Cell RNA雲平台- 線上分析工具
▶ 任你自由調整的篩選門檻與參數 線上調整樣品名稱,更動細胞數篩選及分群的各個門檻
▶ 把圖變成你喜歡的顏色與形狀 直接在平台上調整製圖大小、標題、顏色、字體
提供多種下載格式,製作報告好簡單
▶ 批次效應評估與校正
提供多項套件,修正批次效應影響的細胞分群
▶ 最豐富的分析項目
差異基因分析、基因富集分析(GO、KEGG)、細胞類型標記 (人類及小鼠)
偽時間分析視覺化呈現等,套件持續更新
▶ 深度整合Loupe Cell Browser,讓分析結果最大化
直接匯入差異基因列表,清楚解析基因功能
觀看scRNAseq雲平台直播影片
圖爾思雲平台入口
進階分析項目
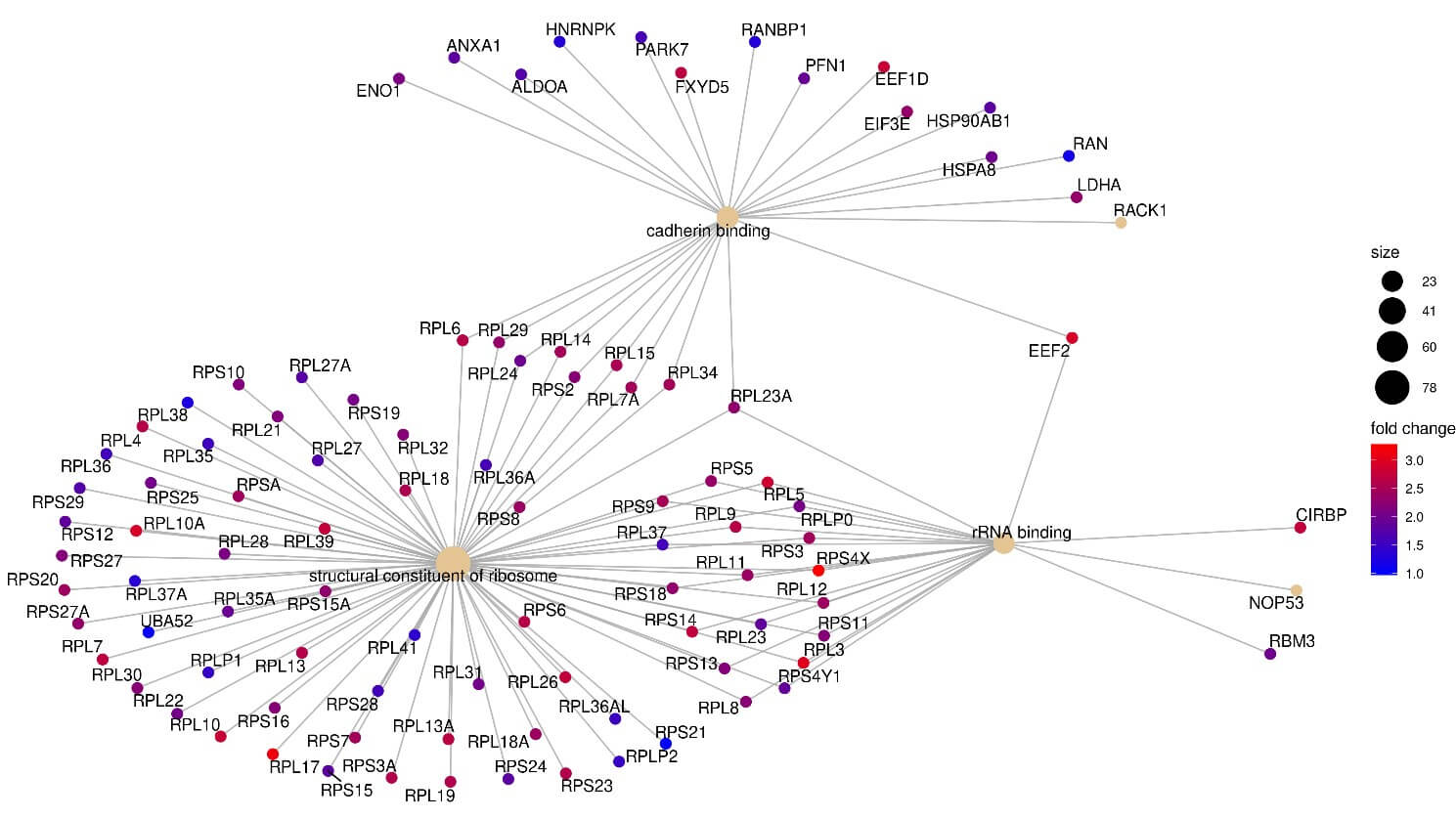
- GO
篩選差異基因,分析差異基因功能路徑。
(* 雲平台匯入Loupe 差異基因計算結果,可匯入第一階段分析不另行收費)
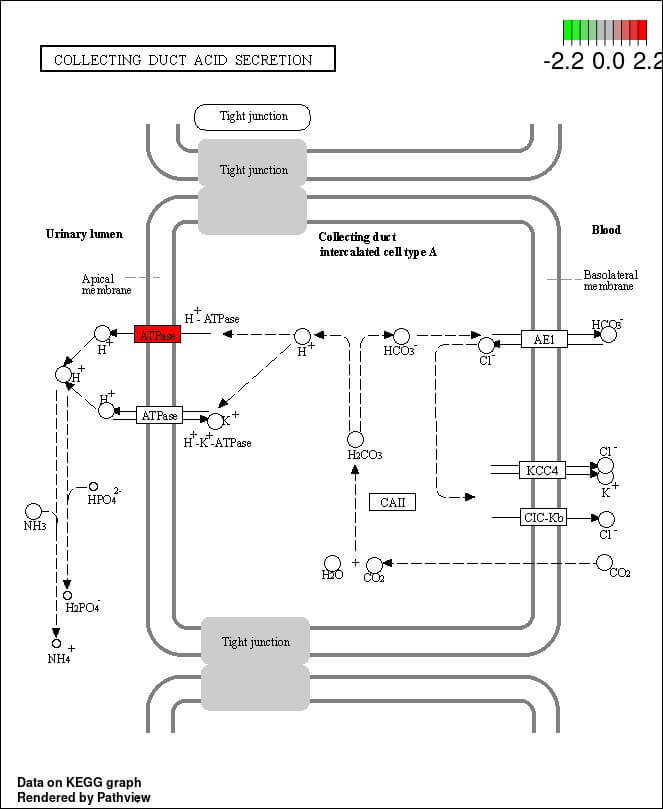
- KEGG
篩選差異基因,分析差異基因參與之代謝路徑。
(* 雲平台匯入Loupe 差異基因計算結果,可匯入第一階段分析不另行收費)
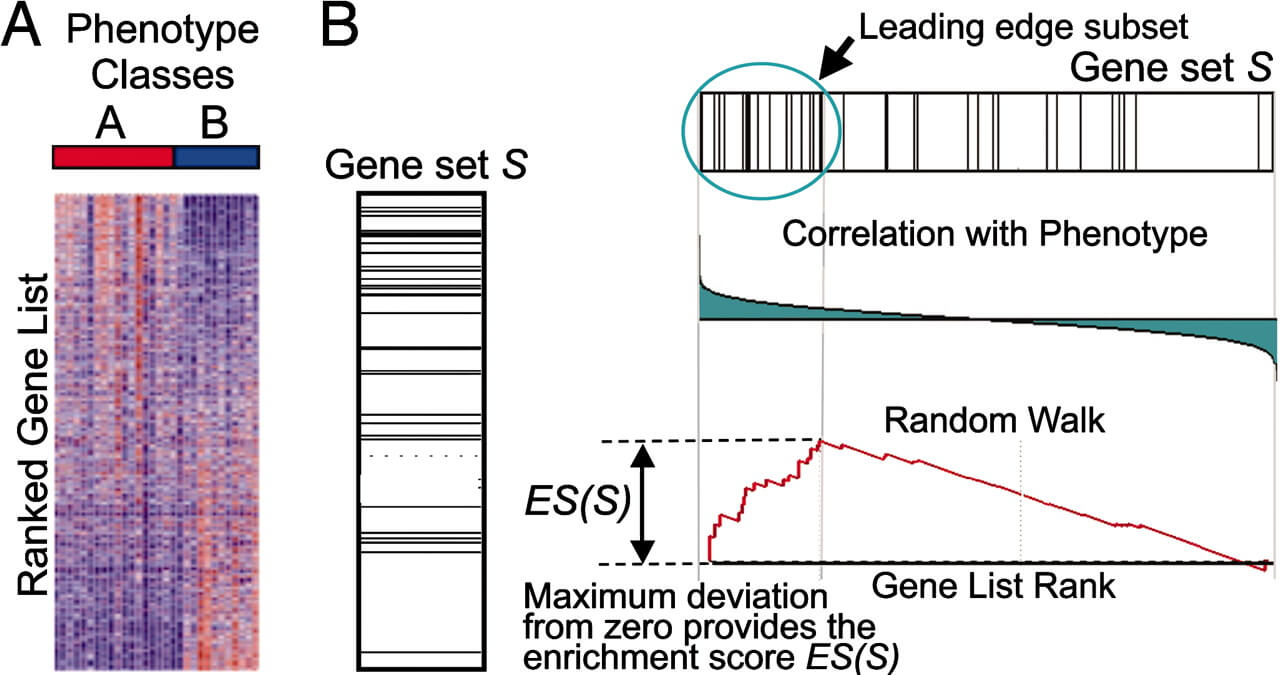
- GSEA
不篩選差異基因,DEG fold-change進行排序後,與預先定義的基因集相比,找出「差異不明顯但趨勢一致」的功能基因集。
(詳見:次世代定序知識櫥窗http://toolsbiotech.blog.fc2.com/blog-entry-118.html)
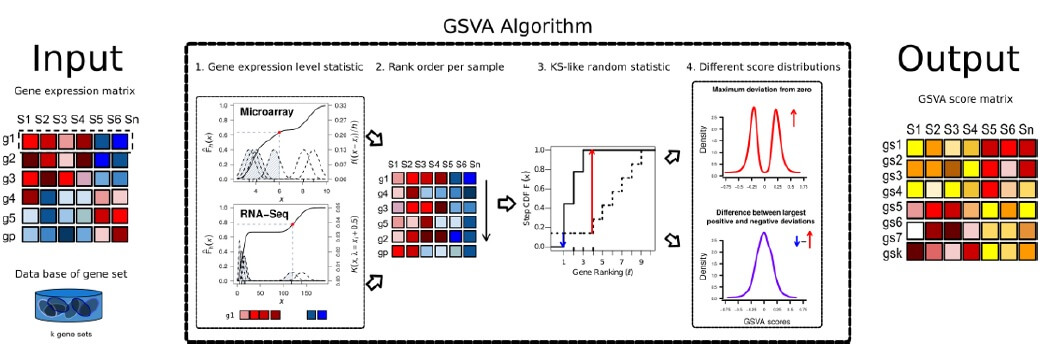
- GSVA
不進行差異基因分析,以單顆細胞為單位對每個基因進行表現量排序,並依照「排序值」、「基因是否在預先定義的基因集內」計算GSVA分數;最後可得「基因集(基因集內所有基因的綜合表現量)-細胞」之分數矩陣。配合tSNE/ UMAP plot,若觀察到一群細胞聚類且GSVA分數高,則意味著此群細胞相比其他細胞群具有此基因集富集。
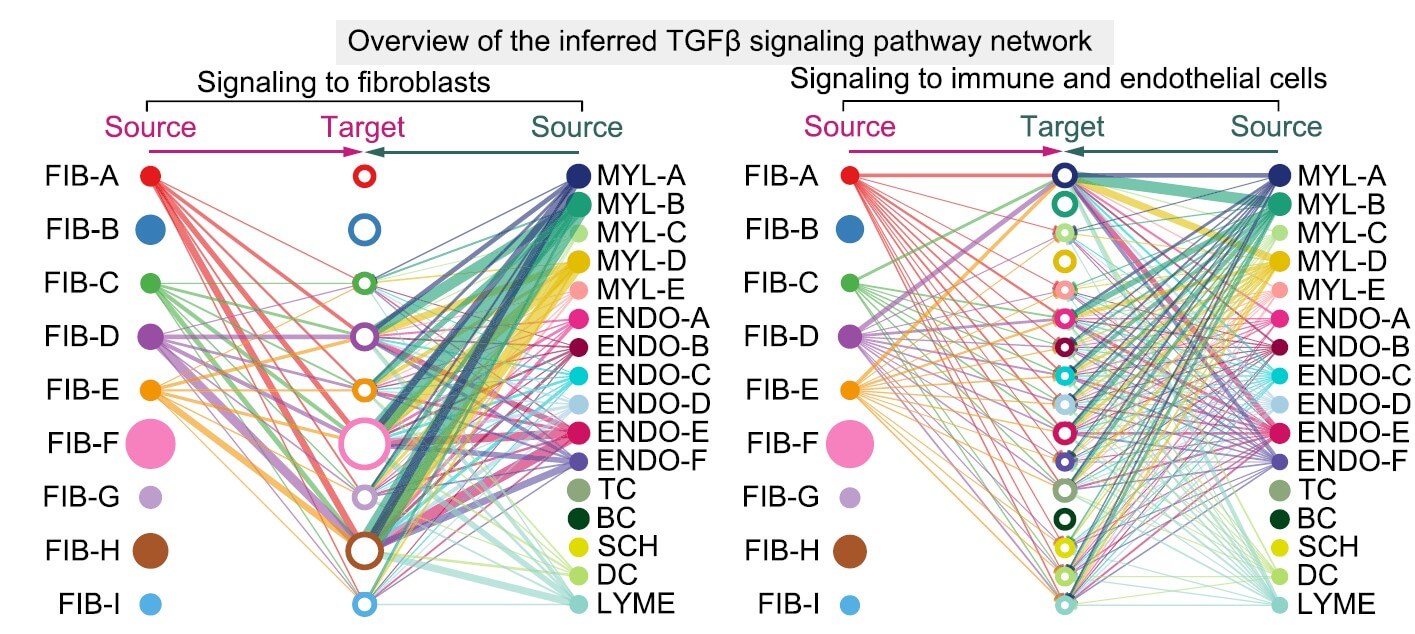
- LR-network
利用Ligand-Receptor判斷細胞群之間的互動性。
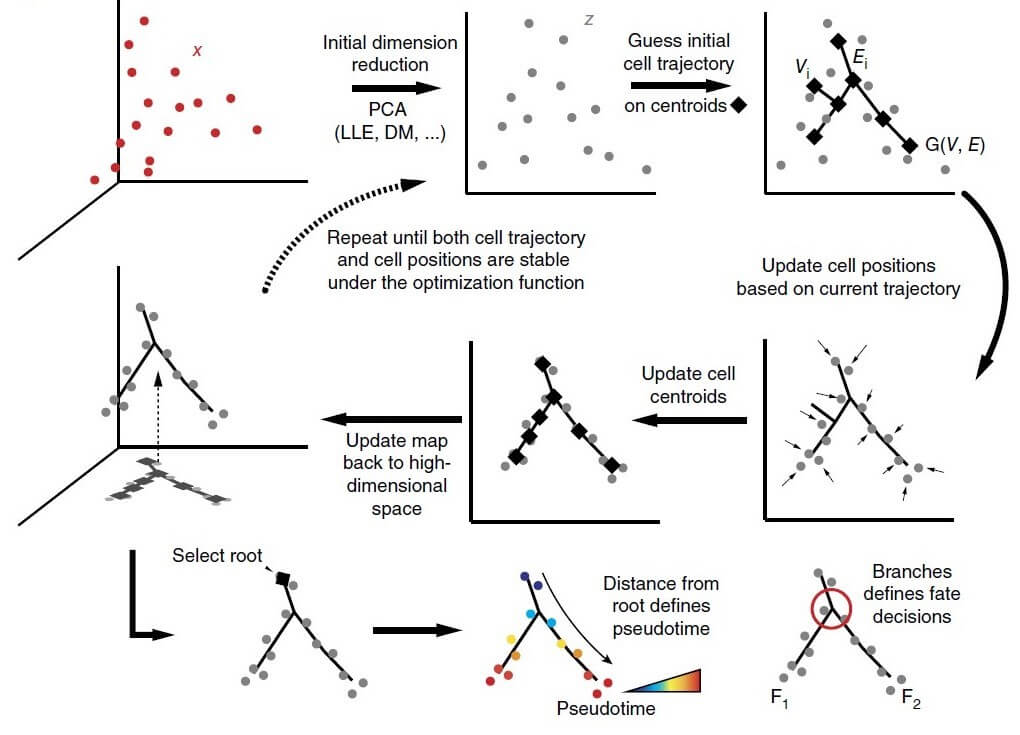
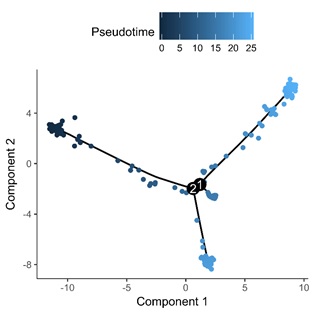
- Trajectory
「模擬」細胞演化之軌跡,輔助推測細胞可能的遷移性。
(* 雲平台提供客製化計算結果之視覺化呈現)
參考資料:
[1] Gene Ontology, C., The Gene Ontology project in 2008. Nucleic acids research, 2008. 36(Database issue): p. D440-D444
[2] Yu, G., et al., clusterProfiler: an R package for comparing biological themes among gene clusters. Omics, 2012. 16(5): p. 284-7.
[3] Kanehisa, M., et al., KEGG for linking genomes to life and the environment. Nucleic Acids Res, 2008. 36(Database issue): p. D480-4.
[4] Kanehisa, M., et al., New approach for understanding genome variations in KEGG. Nucleic Acids Res, 2019. 47(D1): p. D590-d595.
[5] Subramanian, A., et al., Gene set enrichment analysis: A knowledge-based approach for interpreting genome-wide expression profiles. Proceedings of the National Academy of Sciences, 2005. 102(43): p. 15545.
[6] Hänzelmann, S., R. Castelo, and J. Guinney, GSVA: gene set variation analysis for microarray and RNA-Seq data. BMC Bioinformatics, 2013. 14(1): p. 7.
[7] Jin, S., et al., Inference and analysis of cell-cell communication using CellChat. Nature Communications, 2021. 12(1): p. 1088.
[8] Qiu, X., et al., Reversed graph embedding resolves complex single-cell trajectories. Nature Methods, 2017. 14(10): p. 979-982.