解析細胞生理途徑的利器:Perturb-seq
由於Perturb-seq能夠達到高度專一性的基因表現干擾,搭配單一細胞的轉錄體定序,使得上述的高解析度轉錄體變為可能。說起Perturb-seq(見下圖[1]),它的核心概念是CRISPR-interference,意即利用single guide RNA (sgRNA)的專一性,引導不活化的Cas9 (dCas9)到達目標基因,並抑制其轉錄表現。如果目標基因有數十種,那麼便有數十種不同的sgRNA設計,形成一個pooled sgRNA library。利用病毒做為載體,將可表現sgRNA的外源DNA送到細胞內,便可達到目標基因的轉錄抑制。一般而言,在感染濃度低(MOI < 1)的情況下,大多數細胞只會收到一種sgRNA,為單一基因抑制。如果想達成多重基因抑制,可適量提高MOI。
目標基因的轉錄抑制後,為了研究單一細胞整體轉錄的變化,進行單一細胞定序。目前10X Genomics提供的技術可以針對單一細胞的轉錄體產生有條碼標記的cDNA library(見圖[2]的cell barcode),經過次世代定序後,每一個細胞的轉錄體可以透過獨特的條碼序列被辨認出來。
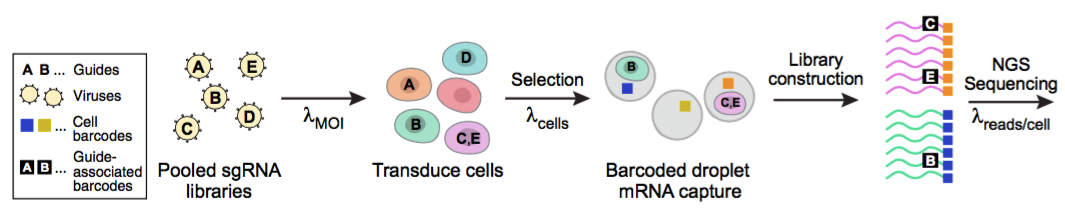
圖[1] Perturb-seq概念
由於每個細胞的轉錄體受到特定sgRNA的影響,下一個重要的步驟是鑑定單一個轉錄體中sgRNA的種類,藉此找出相對應的被抑制的基因。下圖[2]描述的是sgRNA載體的設計,在sgRNA表現區域的下游有另一組用來做為篩選細胞的報告基因(reporter gene),它們帶有一小段guide barcode(GBC),即是用來辨認sgRNA種類的記號。圖中cell barcode是10X Genomics的技術成份之一,不同細胞的轉錄體有不同的cell barcode。
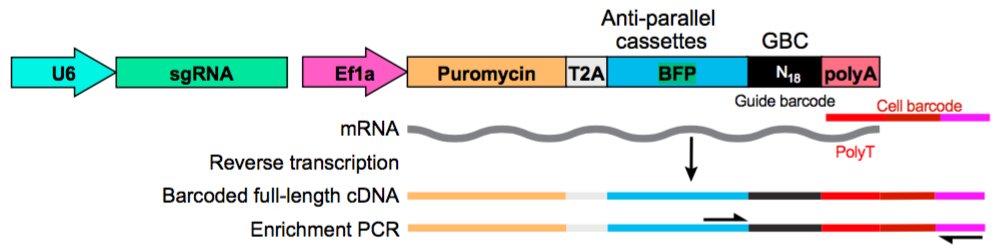
圖[2] Perturb-seq的sgRNA載體
最後小編就來分享一下Perturb-seq的一些成果:圖[3]是一種相關係數熱圖,下方橫軸是不同的sgRNA,對應到最上方的是它們的目標抑制基因(Targeted genes),縱軸是各種受影響的基因,調控係數(regulatory coefficients)的高低以紅色和藍色標示。這張圖可以看出不同的免疫相關基因表現(P1-P5)是由數種轉錄機制(M1-M4)調控的。
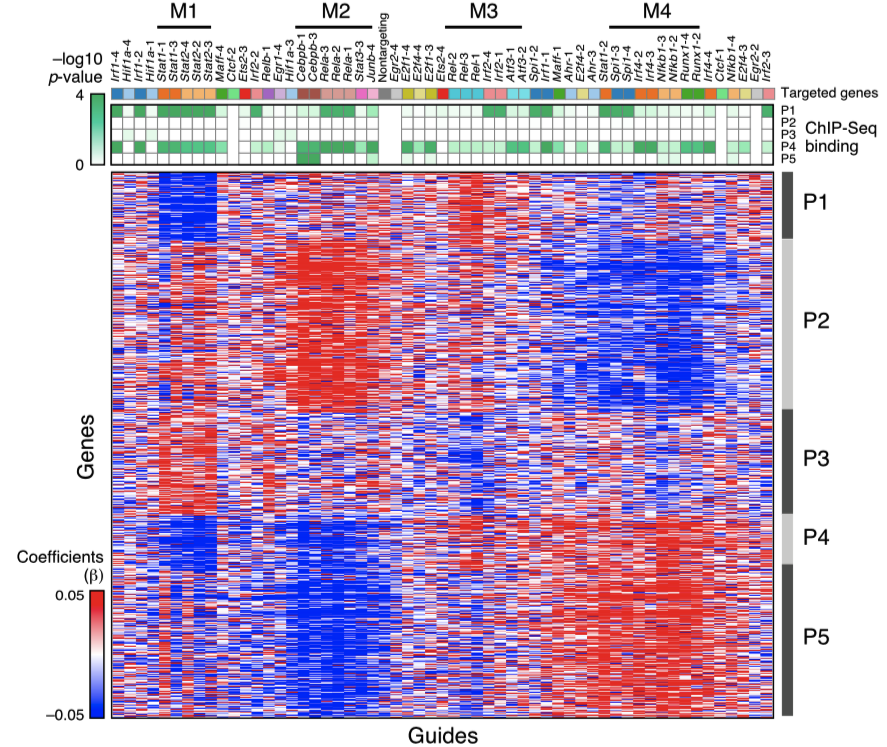
圖[3]以Perturb-seq研究細胞免疫調控相關途徑的結果(參考文獻[1])
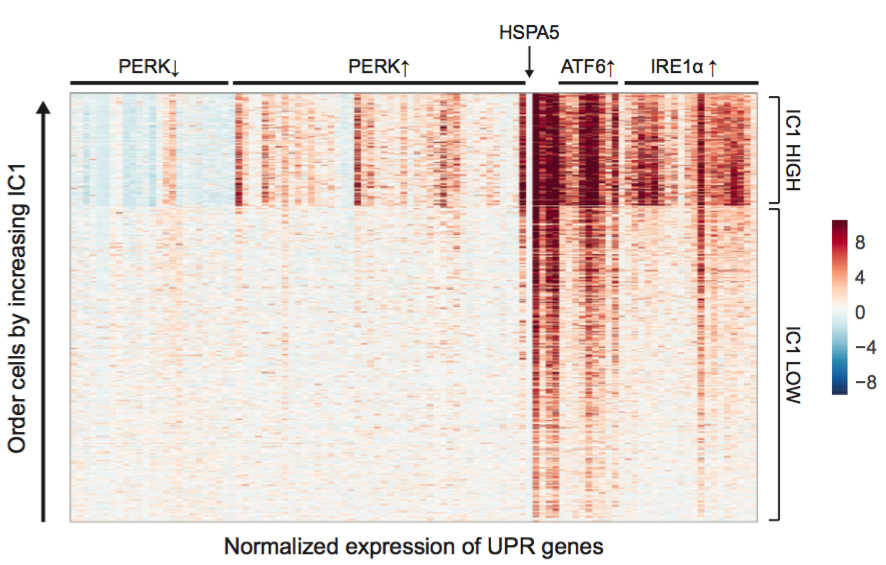
圖[4]是基因轉錄表現熱圖,實驗目的是研究unfolded protein response的三種監測蛋白(sensor proteins)對於下游基因轉錄表現的影響。每一欄是單一種基因在不同細胞的轉錄表現,每一列是單一個細胞的轉錄體。經過聚類分析後,顯示不同的監測蛋白各自影響的下游基因區塊。
搭配客製化的sgRNA library 以及單一細胞轉錄體的定序技術,Perturb-seq 的應用潛力不可限量。這項技術所產生的高解析度轉錄體對於真核細胞pathway 的發掘和建立都有很大的助力,希望有一天能應用在微生物轉錄體的研究上,讓生命科學的世界更多采多姿囉!
參考文獻
[1] Dixit, A., Parnas, O., Li, B., Chen, J., Fulco, CP, Jerby-Arnon, L., et al. (2016). Perturb-Seq: Dissecting Molecular Circuits with Scalable Single-Cell RNA Profiling of Pooled Genetic Screens. Cell, 167 (7), 1853–1857.e17.
[2] Adamson, B., Norman, TM, Jost, M., Cho, MY, Nuñez, JK, Chen, Y., et al. (2016). A Multiplexed Single-Cell CRISPR Screening Platform Enables Systematic Dissection of the Unfolded Protein Response. Cell , 167 (7), 1867–1873.e21.
[3] Jaitin, DA, Weiner, A., Yofe, I., Lara-Astiaso, D., Keren-Shaul, H., David, E., et al. (2016). Dissecting Immune Circuits by Linking CRISPR- Pooled Screens with Single-Cell RNA-Seq. Cell , 167 (7), 1883–1888.e15.