15
2016.10
簡化基因體定序技術-GBS(Genotyping-by-Sequencing)
GBS(Genotyping-by-Sequencing)是一種常見的簡化基因體定序技術,其原理是將基因體DNA進行酶切,然後對酶切片段兩端序列進行高通量定序,透過分析獲得SNP資訊並進行基因分型,是一種快速、簡便、低成本的基因分型方法,適合對大規模樣品進行標記篩選。近年來,透過GBS技術進行高通量定序,發表的文章數量逐年攀升,其中不乏高分文章!接下來小編將帶領大家來領略一下GBS的風采。
案例一 基於GBS技術的高粱農藝性狀全基因體關聯分析
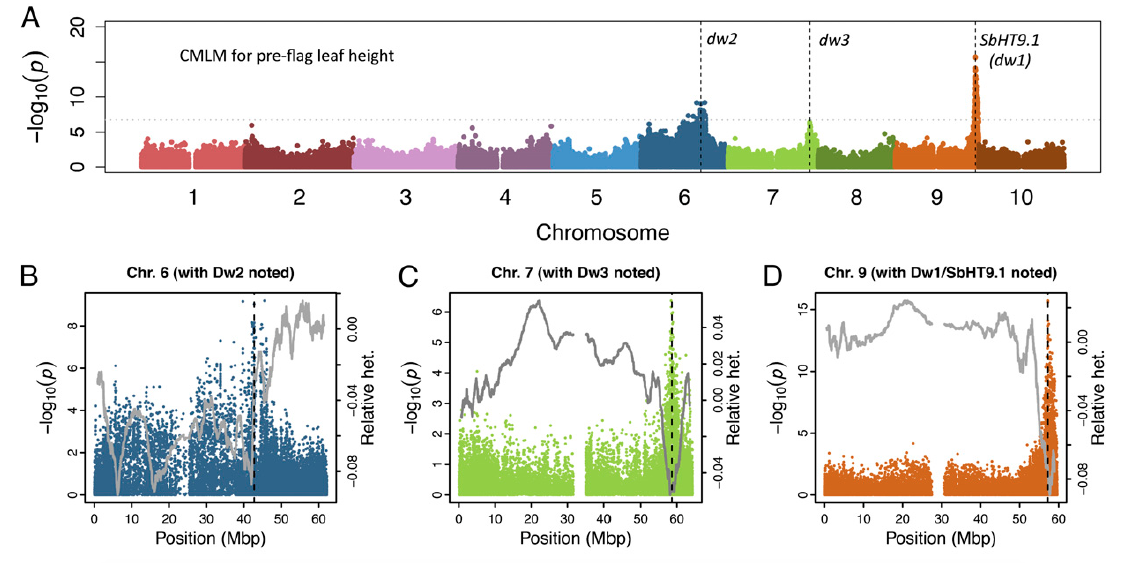
此研究2013年發表在PNAS,研究人員使用GBS技術對來自世界各地的971個高粱品種進行高通量定序,發現了265,000萬個SNP。利用覆蓋全基因體的SNP圖譜,研究者描述了高粱地理起源的群體結構、形態特徵,並確認出祖先穀物向多樣性農業氣候的亞非地區擴散的模式。此外,研究人員透過全基因體關聯分析(GWAS)研究株高性狀和花序結構。基於群體分層分析結果,將971份高粱品系中的336份品系用於高粱株高的全基因體關聯分析。透過GWAS發現,Dw3與前旗葉高度相關,Dw1、Dw2和Dw4均與頂端旗葉高度相關,Dw3和Dw2與整體株高相關。該研究找到了與高粱株高相關的已驗證位點以及花序結構性狀相關的候選位點,為分子輔助育種和基因體選擇改善穀物提供了基礎。
圖1. 高粱全基因體關聯分析
案列二 基於GBS技術構建遺傳圖譜,結合GWAS進行高粱株高性狀定位
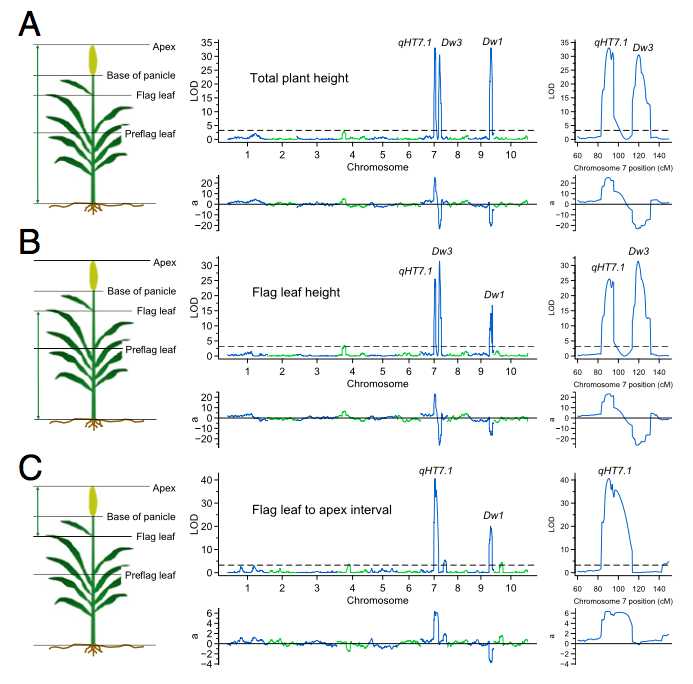
此研究2015年發表在PNAS。研究人員透過Tx430 × P898012構建了一個高粱的RIL群體,群體大小為250個,且RIL群體的株高符合正態分佈。透過GBS定序手段共獲得8,960個SNP,其中1,756個標記用於構建遺傳圖譜,遺傳距離總長為1916cM,標記間平均遺傳距離為1.3cM。透過QTL定位,共檢測到3個QTL與株高相關(Dw1 , Dw3和qHT7.1),分別位於第7號染色體和第9號染色體上。在第7號染色體上檢測到了2個QTL,且在這兩個QTL附近均可以檢測到Dw3基因。對位於第9號染色體上的QTL區間內進行功能註釋,經分析發現Dw1位於該區間內。透過分析Dw3發現,Dw3等位基因在Tx430的第五個外顯子區域有882bp的缺失。
圖2. 高粱QTL定位
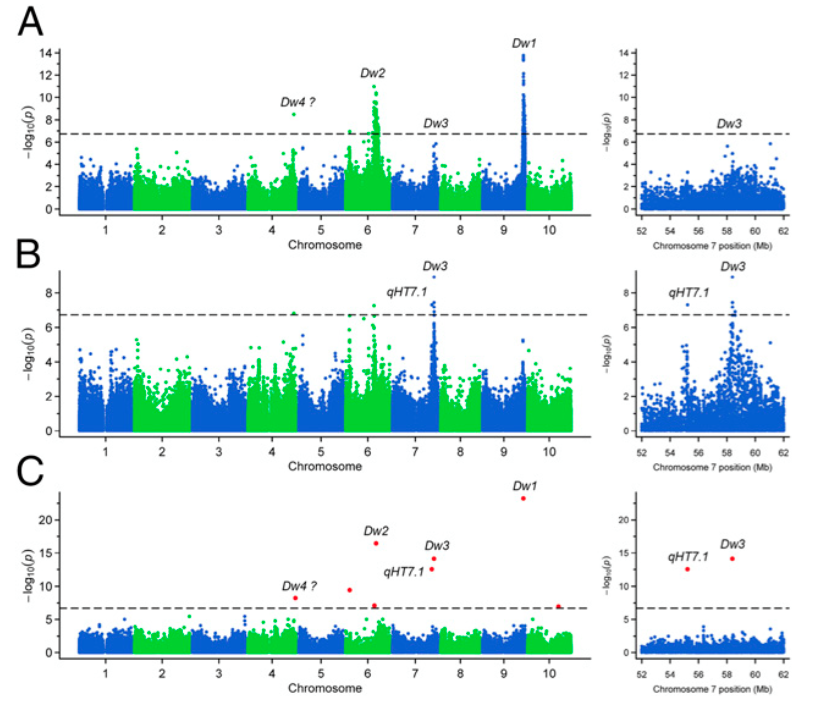
隨後,利用2013年發表在PNAS上相同的群體進行全基因體關聯分析,其中群體中378個個體的基因型資訊為重複利用的數據,透過GWAS的多種混合模型的計算,挖掘了到了qHT7.1,說明qHT7.1是控制株高的主效QTL。此研究結合連鎖分析和關聯分析的優點,分別從縱向和橫向對高粱株高性狀進行剖析,加速了目標性狀基因的鑑定,為深入認識高粱株高性狀的遺傳學和分子生物學基礎提供了一個方向。
圖3. 高粱全基因體關聯分析
案例三 基於GBS技術研究鷹嘴豆的遺傳多樣性
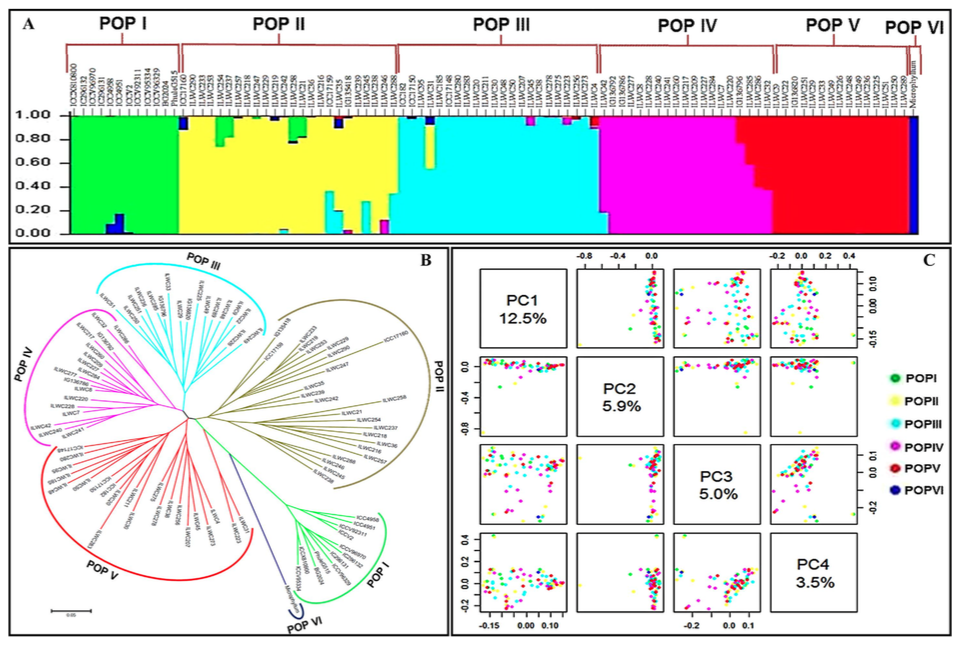
研究人員對93株鷹嘴豆(包括栽培種和野生種)利用GBS技術進行高通量定序,定序平台為Illumima HiSeq 2000,PE100 ,平均定序深度為20X。經群體遺傳結構、群體進化樹和主成分分析,都將93株鷹嘴豆分為6個亞群(POP I-POP VI),三種分析方法的結果相互驗證,說明這是最理想的群體分類結果。隨後對這些亞群進行連鎖不平衡分析,結果顯示不同的群體在進化過程中受選擇程度不同,其中POPIII的LD值最小,群體POPI的LD值最大,說明POPI受選擇的程度最大,遺傳多樣性最低。該研究透過GBS技術對鷹嘴豆進行遺傳多樣性研究,為重要經濟性狀新基因的挖掘及基因滲入等研究奠定了基礎。
圖4. 鷹嘴豆群體遺傳多樣性分析
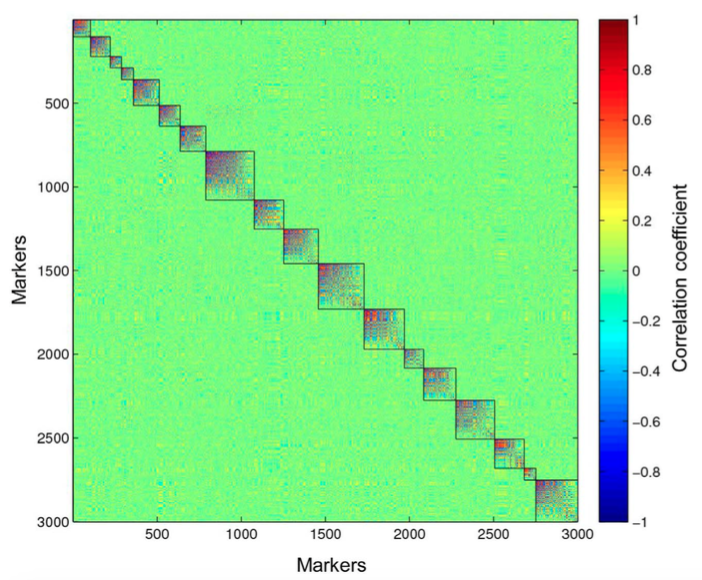
案例四 基於GBS技術研究柳枝稷遺傳多態性、倍性及進化
研究人員對兩類柳枝稷(Switchgrass)群體,分別是關聯panel(540個)和連鎖群體(130株F 1全同胞家系,親本為四倍體柳枝稷U518和U418;168株半同胞家系),利用Illumina Hiseq 2000平台對這840個柳枝稷個體進行GBS定序,共產生了350Gb資料。結合UNEAK方法在主要高地和北方適應性柳枝稷群體中開發出120萬個潛在的SNPs,對這些數據進一步分析發現,四倍體柳枝稷展現出了重要的二倍體特性。結合柳枝稷和穀子基因體結構高保守性優勢及共線性而構建了超高密度的遺傳連鎖圖譜(包含有88,217 SNPs)。系統進化分析證明了柳枝稷從南至北的進化遷移,每種生態型分佈於不同的地理位置,高地四倍體柳枝稷是來自於高地八倍體。
圖5. 柳枝稷父本18條連鎖群的熱圖分析
此研究利用GBS並結合適合於高雜合複雜基因體物種的UNEAK方法對柳枝稷的遺傳多樣性進行了深入系統研究,主要包括多樣性、遺傳複雜性、群體結構、種係發生、地理分佈、倍性和進化動力學等,首次展現了柳枝稷的複雜遺傳多樣性研究結果。
參考文獻
[1] Morris GP, Ramu P, Deshpande SP, et al . Population genomic and genome-wide association studies of agroclimatic traits in sorghum[J]. Proceedings of the National Academy of Sciences , 2013, 110(2): 453-458 .
[2] Li X, Li X, Fridman E, et al . Dissecting repulsion linkage in the dwarfing gene Dw3 regionfor sorghum plant height provides insights into heterosis.[J]. Proceedings ofthe National Academy of Sciences of the United States of America , 2015 ,112(38):11823-8.
[3] Bajaj D, Das S, Badoni S, et al . Genome-wide high-throughput SNP discovery and genotyping for understanding natural (functional) allelic diversity and domestication patterns in wild chickpea[J]. Scientific Reports , 2015, 5.
[4] Lu F, Lipka AE, Glaubitz J, et al . Switchgrass Genomic Diversity, Ploidy, and Evolution: Novel Insights from a Network-Based SNP Discovery Protocol[J]. Plos Genetics , 2013, 9(1):139- 147.