19
2016.05
RNA定序策略選擇的糾結!
各位在NGS-轉錄組研究沙場上拼鬥時,是否曾糾結過定序策略的選擇問題? 糾結1. 要進行雙端 (Paired End) 定序,還是單端 (Single End) 已足夠? 糾結2. 讀長 (read) 的選擇又會如何影響後期的差異基因篩選? 糾結3. 定序讀長的選擇是否越長越好?
這些糾結問題,我們就交由一篇發表於 Genome Biology 的文章來面對大家好好解釋交代一番~~
該文作者藉由對雙端101bp定序序列截取生成25bp、50bp、75bp及100bp的單端或雙端序列,進行表現差異分析及剪接體檢測,發現針對不同研究目的,不同定序策略得到的結果可真是令人驚訝~~~
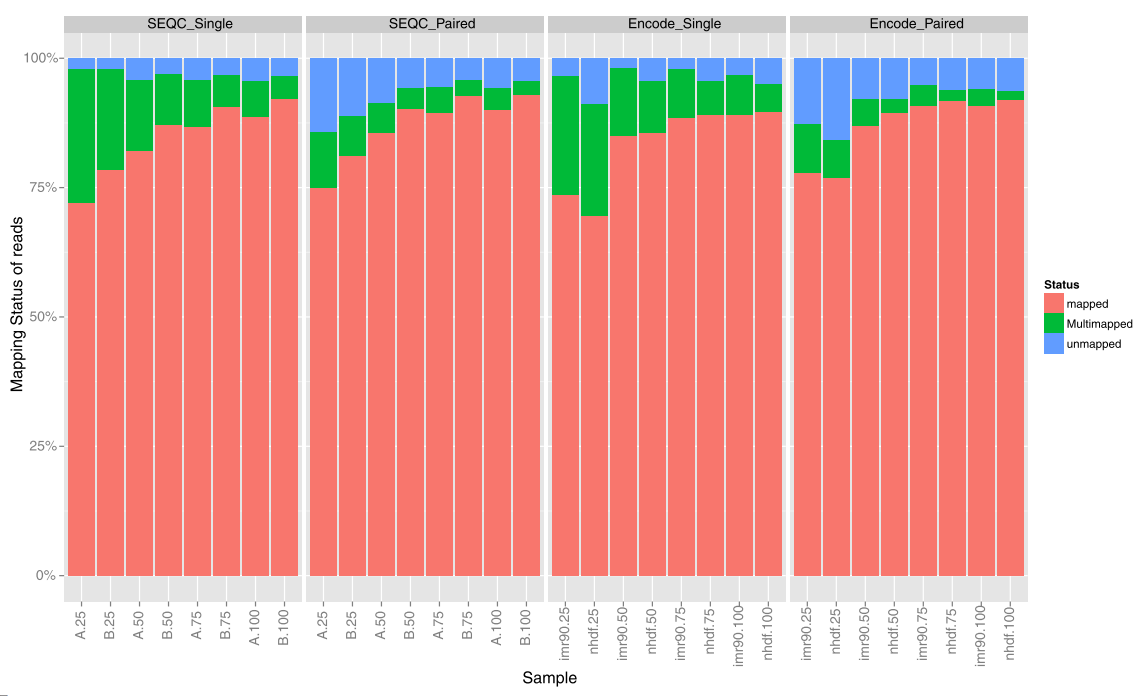
1. 定序策略對 mapping 率的影響 25bp序列單一mapping率最差,隨著序列長度的增加mapping率隨之提高,而75bp和100bp序列有著幾乎一致的單一mapping率。其中,單端25bp序列下的multi-mapping率相較於單一mapping率有顯著提高,而單端策略下的50bp、75bp和100bp序列有著近於一致的multi-mapping率。
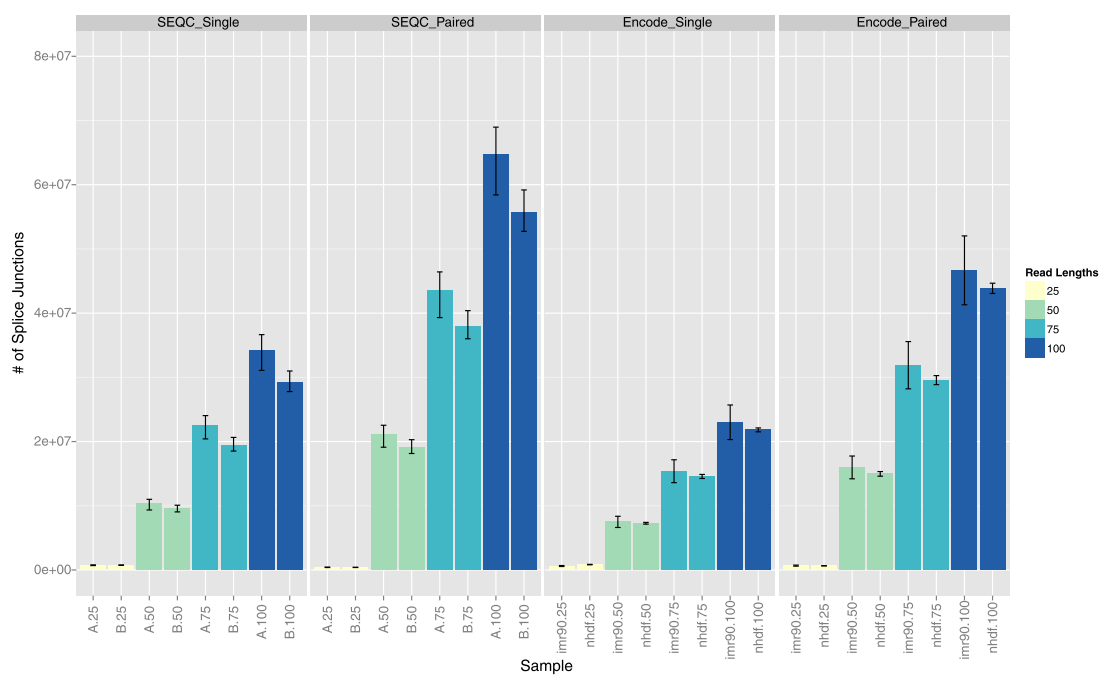
2. 定序策略對剪接體檢測的影響
在剪接體檢測方面,25bp讀長表現最差,隨著定序長度的增加,研究人員檢測到了更多的剪接體。同時,雙端策略顯著優於單端策略。
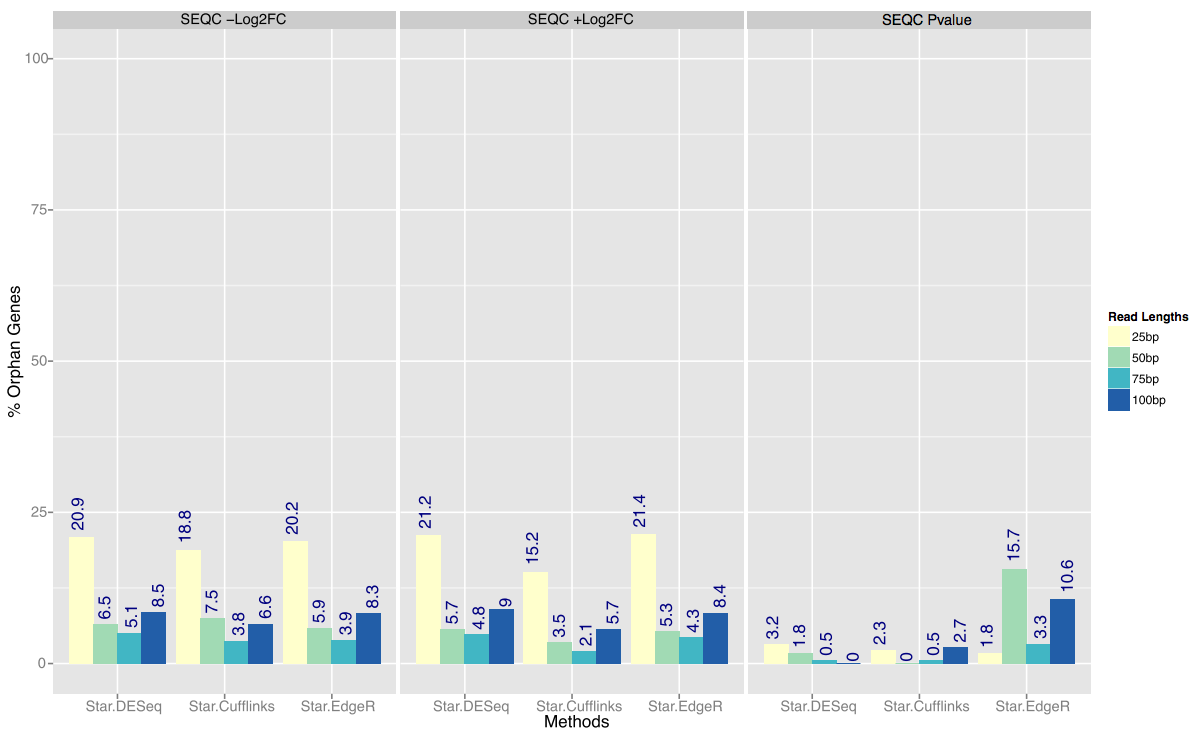
3. 定序策略對差異基因篩選的影響
研究人員利用三種套件(DESeq、EdgeR和Cufflinks)分別篩選出前200個表現量有顯著差異的基因。發現,單端25bp讀長策略下,孤兒基因(只能在特定讀長中被檢出的基因)數目的比例是最大的,也就是說25bp讀長在篩選差異基因方面和其他策略顯著不同。雖然改為雙端策略可以削弱這種影響,但雙端策略下25bp讀長仍是最差的選擇。而在50bp、75bp和100bp讀長下,無論單端還是雙端策略,孤兒基因的檢出水平都相近。
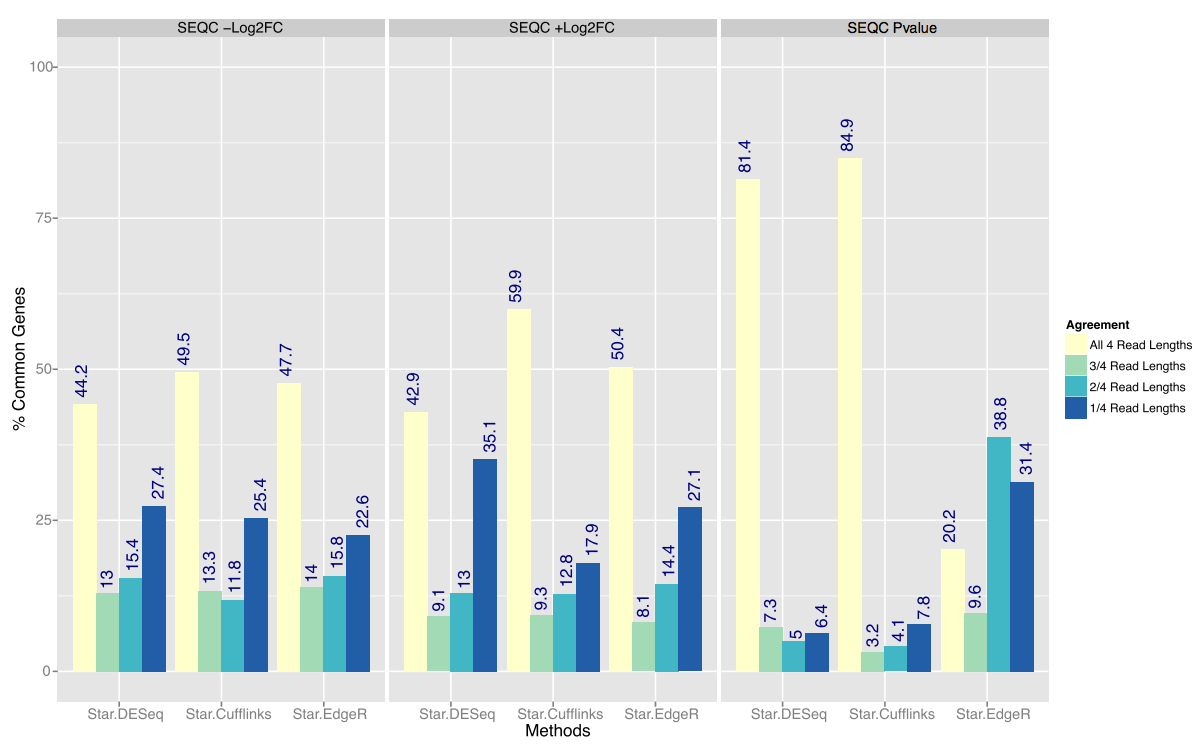
而對於四種讀長下都能檢出的差異基因,即使在同等讀長下,雙端策略也顯著優於單端策略。
最後,研究人員用qPCR實驗驗證,對於差異基因的檢測,雙端策略下75bp和100bp讀長結果相似,而單端策略下,100bp讀長產生最好的結果。
縱上所述,RNA-seq中,定序策略最好選擇 > 50bp。如果要進行比對分析和剪接突變體檢測,雙端測序有比較顯著的優勢。但對於差異基因分析,選用雙端定序效果可能並不會明顯優於單端定序。
圖爾思生物科技 / 諾禾致源文案