培養皿中的腸道居民
原創文章 引用請註明出處
說起人類腸道微生物 ,
以往的觀念是牠們喜歡養分充足、溫暖而缺乏氧氣的環境,而且大多數無法在實驗室條件下培養成功。今年(2016) Trevor Lawley博士的研究團隊發表在Nature的一篇文章,卻推翻了人們對腸道菌的既定印象。文章中提到的幾個有趣的小發現,小編整理出來和大家分享:
(1) 大多數腸道菌是可以在適當的實驗條件下培養的,而且只需要三天
(2) 約有一半的腸道菌種可以形成孢子,有利於在人類之間傳播
(3) 經過一段時間後,腸道中可形成孢子的細菌比不形成孢子的細菌,數量和種類的變化更劇烈
除了以上幾點,他們的研究團隊更是找出了90種美國Human Microbiome Project (HMP)最想要定序卻苦於沒有材料(就是細菌本身啦~)的菌種,因為過去大家都認為絕大多數的腸道菌是無法培養的。Lawley博士的團隊不只鑑定出以前沒有被大家發現的細菌,還把這些細菌冷凍保種,以後如果要進一步認識這些新種腸道菌,就不用再辛苦地收集人類大便了,去冷凍庫挖就有,是不是很棒呢 ?想當然爾,對大量的菌種做鑑定、分離和保存是非常花工夫的,光是鑑定一個菌種就要針對全長16S核糖體基因做定序。話不多說,接下來就讓我們一起來好好欣賞研究團隊的成果吧~
第一階段:腸道菌相的培養、分離和鑑定
最重要的第一步,就是收集健康人類的糞便並且試圖培養其中的腸道菌種。該團隊收集了六位不同健康受試者的新鮮糞便樣本,一部分馬上定序以鑑定菌種,另一部分則是以YCFA固態培養基在無氧環境下嘗試培養其中的腸道菌。
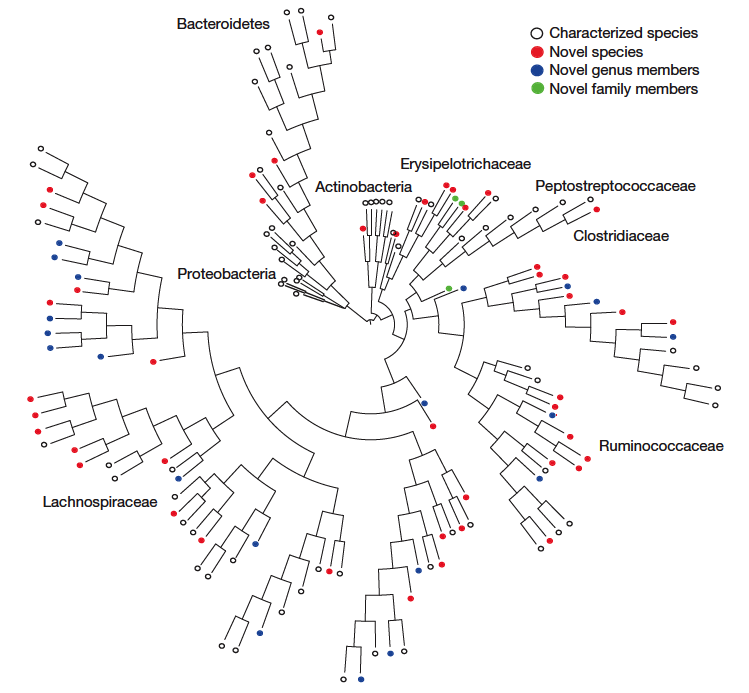
為了找出可能產生孢子的菌種,他們將培養後的細菌們以酒精處理,殺死那些不會產生孢子的細菌種類(因為孢子對酒精有抵抗力,所以這個步驟可以去除不會產生孢子的細菌),再次培養後的菌相和原本腸道菌相比對照下,就可以找出哪些是有可能產生孢子的細菌囉。在YCFA上培養的腸道菌,其菌相組成幾乎可以代表糞便樣本的菌相[圖1],而酒精處理後的培養菌相變化,很明顯地讓我們知道原來腸道菌中很大一部分的細菌是可以產生孢子的[圖2]。經過菌種鑑定後,研究團隊做了演化分析,讓我們知道新種腸道菌彼此間的親疏遠近[圖3]。
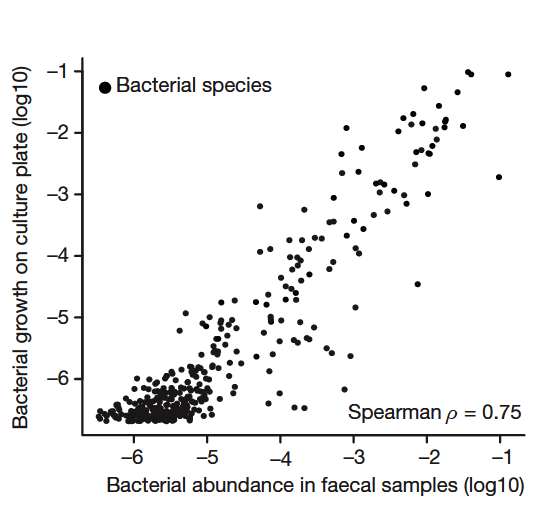
圖1.
人類新鮮糞便(橫軸)和培養後的腸道菌(縱軸)的菌種豐富度的比對
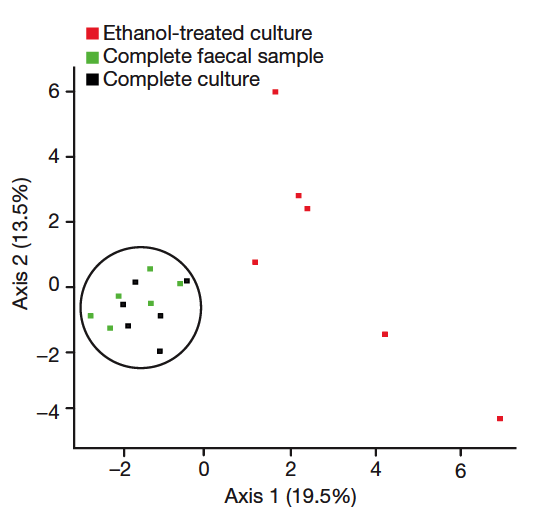
圖2. PCA圖表的三個樣本:酒精處理後的培養菌相(紅點)、未處理的培養菌相(黑點)以及糞便菌相(綠點)
圖3. 新種腸道菌演化分類樹:菌種(紅點)、菌屬(藍點)和菌科(綠點)
第二階段: 針對產生孢子的細菌做分類
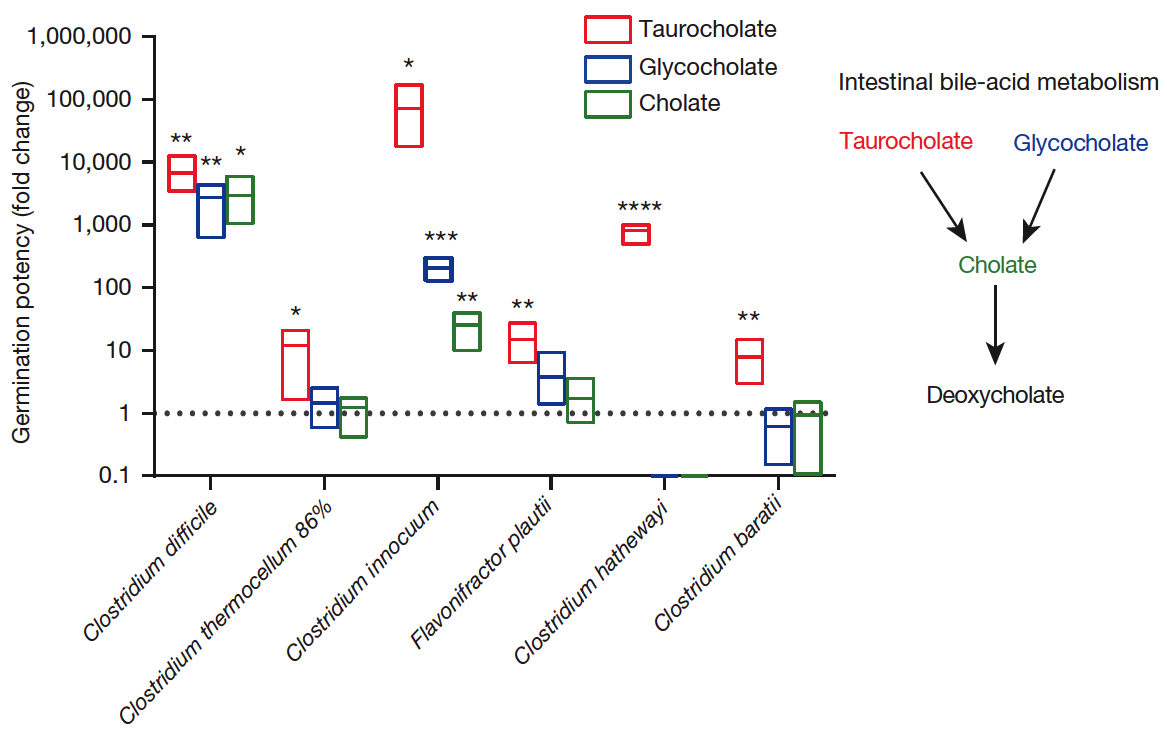
經過第一階段的實驗,產生孢子的細菌種類已經呼之欲出了。下一步就是要詳細地分析牠們對於氧氣的耐受性(別忘了,多數腸道菌是不喜歡氧氣的,而細菌孢子對於氧氣和各種化學物質有高度的抵抗力)以及種類。他們發現不產生孢子的細菌,在一般大氣環境下可以存活2-6天,而產生孢子的細菌,比如困難梭狀桿菌,則可以撐到21天[圖4]。要注意的是存活狀況最佳的大腸桿菌(圖4上方黑色虛線),並不會產生孢子,牠是一種兼氣性細菌,無論有無氧氣都可以活得很開心。為了讓細菌孢子的萌發效率更好,他們特別研究哪種形式的膽鹽對孢子有最好的萌發率,結論是taur-ocholate的效果最明顯[圖5]。
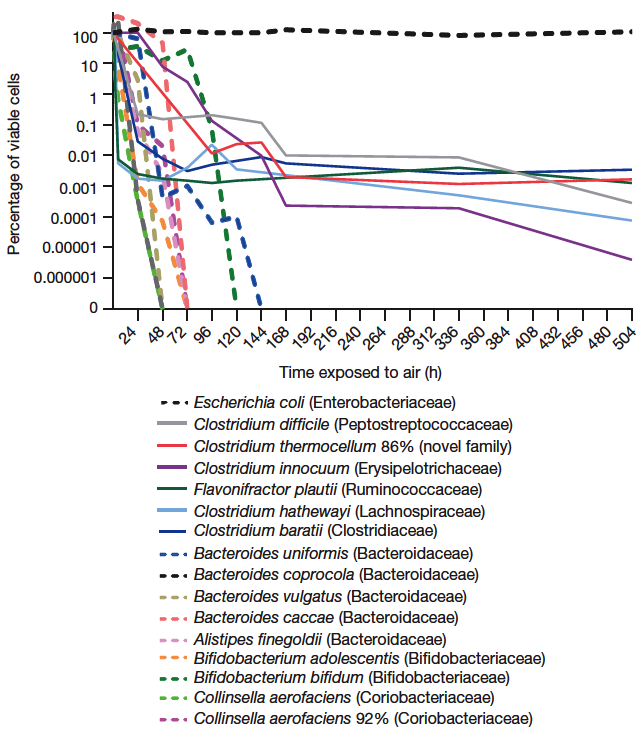
圖4. 橫軸是一般大氣條件下的存活時數,縱軸是存活百分比。虛線是不產生孢子的細菌,實線是可產生孢子的細菌。
圖5. 橫軸是細菌種類,縱軸是萌發效率。
第三階段: 找出有能力產生孢子的細菌在腸道菌中的比例
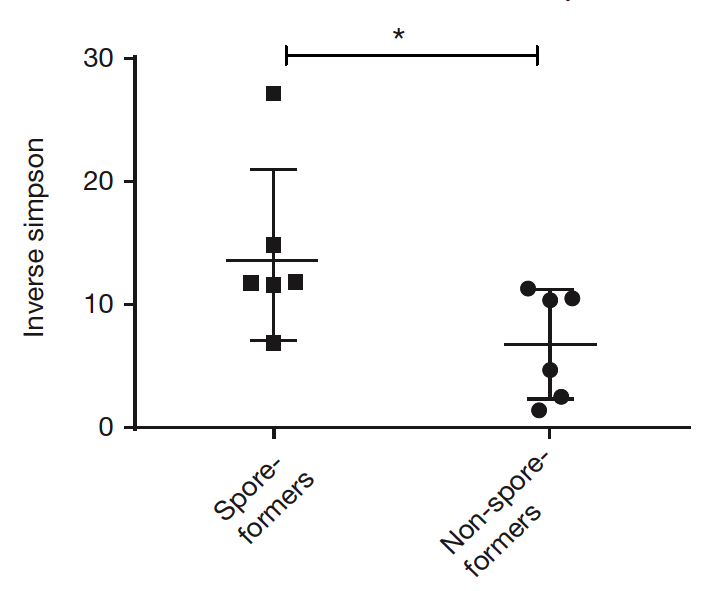
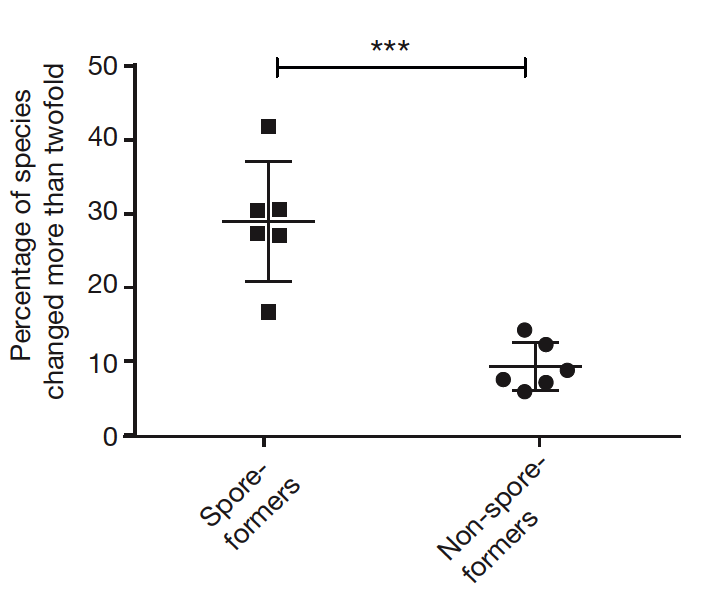
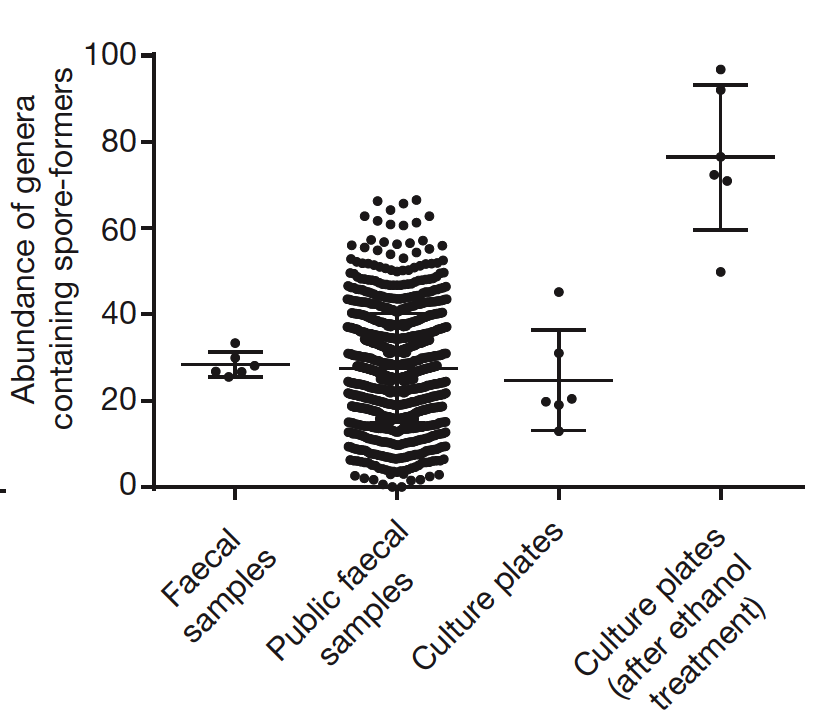
既然我們知道了腸道中某些細菌可以產生孢子,接下來的問題就是:這些細菌在腸道中佔有多少比例?由於次世代定序和生物資訊分析的大幅進展,研究團隊將產生孢子的菌種分類整理後,和開放資料庫裡的人類腸道菌相資料做比對,發現健康人類的腸道菌裡,有50-60%是可以產生孢子的[圖6],而且大約30%的腸道菌屬,含有產孢子細菌[圖7]。除此之外,他們還發現可以產生孢子的菌種的豐富度比不產生孢子的菌種更高[圖8]。隨著時間的推移,可產生孢子的細菌豐富度變化將近30%,而不產生孢子的細菌豐富度變動只有大約10%[圖9]。
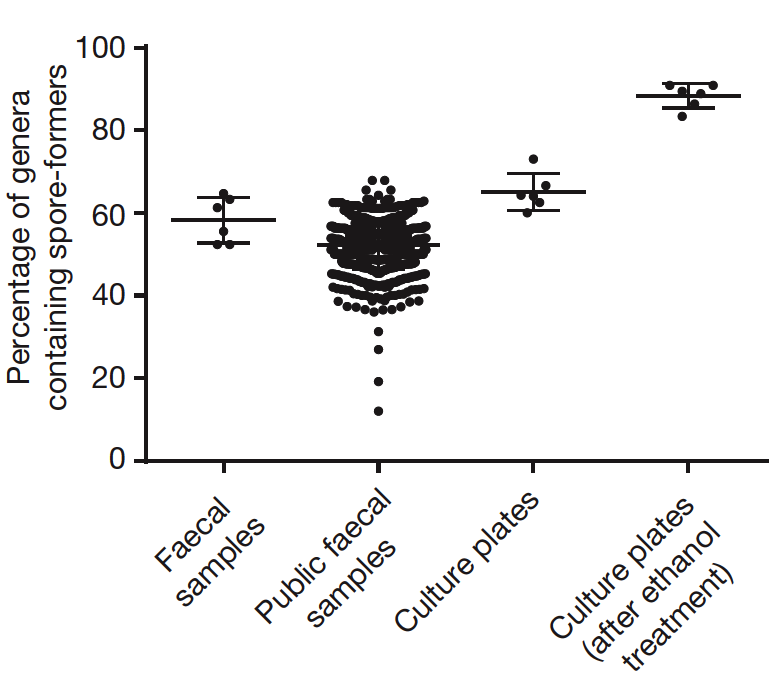
圖6. 橫軸是樣本種類,從左到右依次為:六位受試者樣本、公用資料庫樣本、實驗條件培養腸道菌樣本,以及酒精處理後的腸道菌培養樣本。縱軸顯示的是產孢子細菌在菌屬中所佔比例。
總結
Lawley博士的研究對於未來腸道微生物的培養和分析有莫大貢獻,除了提供腸道菌群移植(參考文章: 精準微生物醫療的新時代 - 糞菌移植)的培養條件之外,科學家們還可以試著改良這些新種腸道居民的基因,讓牠們能夠治療疾病、改善人類體質並且安全地定居在腸道裡。由於腸道菌群移植不像器官移植有排斥的疑慮,小編正期待著菌群移植蔚為流行的那一天呢!
參考文獻
[1] Browne, H. P., Forster, S. C., Anonye, B. O., Kumar, N., Neville, B. A., Stares, M. D., et al. (2016). Culturing of “unculturable” human microbiota reveals novel taxa and extensive sporulation. Nature, 533(7604), 543–546.
圖爾思生物科技/ NGS事業部
許方瑜 文案