【Metagenome小學堂】 誰是微生物探查的最佳解?
原創文章,引用請註明出處
||一首歌的時間,快速了解Metagenome樣品與數據品質的重要性||
Metagenome樣品製備:親手建立屬於微生物們的舞台
shotgun Metagenome—這不是一把真正的霰彈槍,而是一種能夠讓你深入微生物世界的奇妙工具。這種技術讓我們能夠不經培養,直接從環境樣本中萃取DNA進行定序,揭示出那些藏在微小生物體內的基因秘密。
1. 樣品來源:Metagenome研究主要針對微生物群落進行分析,因此樣品來源通常包括環境樣品(如土壤、水體、沉積物等)、人體或動物體內樣品(如腸道內容物、口腔拭子、糞便等)。選擇樣品時,應考慮其代表性和目標微生物群落的多樣性,以確保獲得全面和有意義的數據。
2. DNA萃取:從樣品中萃取DNA是Metagenome研究的重要步驟。我們建議客戶自己萃取DNA,這樣可以確保在樣品處理過程中最大限度地控制變因,並且在樣品處理和運輸過程中減少可能的降解風險。此外,自行萃取還能讓客戶更好地了解並掌控樣品處理的每個步驟,從而提高數據的可靠性。
3. 收樣標準:
DNA 樣品總量 ≥ 1.0 μg (Qubit® 測定)
DNA 樣品濃度 ≥ 50 ng/μL (Qubit® 測定)
樣品體積 ≥ 20 μL
樣品QC時常常會遇到各種的狀況:
(A)DNA Marker, (B) 合格樣品 (C) 降解樣品 (D) RNA汙染樣品 (E) 蛋白質汙染樣品(以紅框標記汙染處)

4. 為何需要完整無降解的DNA?不是都需要進行片段化後才進行建庫嗎?
雖然NGS建庫過程中會進行片段化,但一開始DNA的完整性仍然非常重要,如果DNA在萃取過程中已經嚴重降解,可能會造成下列的問題:
Size Selection的有限性:size selection的效率不是100%的,可能仍然會有一些過小的片段進入定序,造成數據掠奪。
降解程度不均勻:如果DNA樣品在不同區域降解程度不均,導致某些gene無法產出完整的定序結果。
數據可靠性: 嚴重降解的DNA可能影響最終的定序數據品質,導致數據偏差或無法進行有效組裝基因組。
數據QC:微生物數據的美容師
數據品質控制(QC)就像是一位專業的美容師,確保所有的數據都是高品質,沒有Adapter的雜毛,也沒有宿主基因體汙染。這個過程保證數據的準確度和可靠性,讓後續的生物資訊分析就像是在微生物的臉上塗上了完美的妝容,大放其彩。
1. Trimming:使用Trimming軟體對原始數據進行修剪,去除Adapter序列,並去除起始和結尾處的低準確度序列,也會對序列進行長度篩選,去除過短的序列,確保後續分析的準確性和可靠性。
2. Alignment軟體比對宿主基因體:使用Alignment軟體將修剪後的序列比對到人和小鼠的宿主基因體進行宿主序列的去除(根據樣本來源,選擇對應參考基因體),去除完的數據才是可用於微生物分析的美麗資訊。如果宿主DNA汙染程度嚴重,可用於後續分析的數據量就會很低。
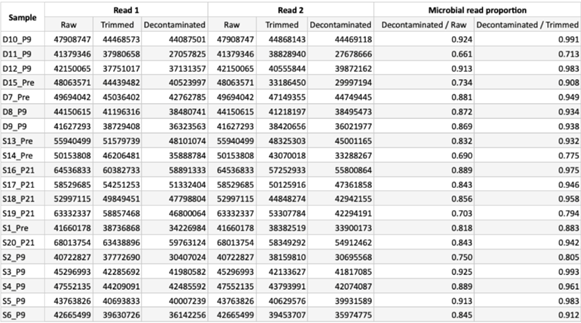
3. 數據QC的統計和視覺化:
<序列數量柱狀圖>能夠觀察到每個樣品的序列數量,可以讓我們評斷樣品序列量多寡。

<定序資料品質表>此分析表提供我們判斷定序深度是否符合預期,與序列數量柱狀圖能一起做為數據是否要進行加測的依據。
卓庭光 文案 http://www.toolsbiotech.com/ © BIOTOOLS. All Rights Reserved



