27
2021.12
【三代定序 新知分享】真菌定序平台比較
【三代定序 新知分享】真菌定序平台比較
細菌定序常見區域為核糖體RNA的16S,真菌定序也有其特定常用區域,通常為核糖體RNA的18S、28S、轉錄間隔區 (Internal Transcribed Spacer, ITS)。ITS區域長度為500-700 bp,可分為ITS1跟ITS2區域,不同真菌之間ITS2長度上的差異較小、有較多通用的primer位點,使用ITS2做為真菌分類上的指標產生的偏差較ITS1少,因此較多真菌高通量研究使用ITS2作為指標區域。而今天要介紹的文獻則以ITS1區域作為定序標的,比較二代定序IonPGM、Miseq和三代定序PacBio定序真菌mock community的結果。
作者使用了15屬、共26種真菌的標準品做正確答案,定序其ITS1區域作為鑑定標準。混和物內含的真菌種類標示如表一,包含人體中常見真菌Candida glabrata、食物製品中的Saccharomyces cerevisiae。

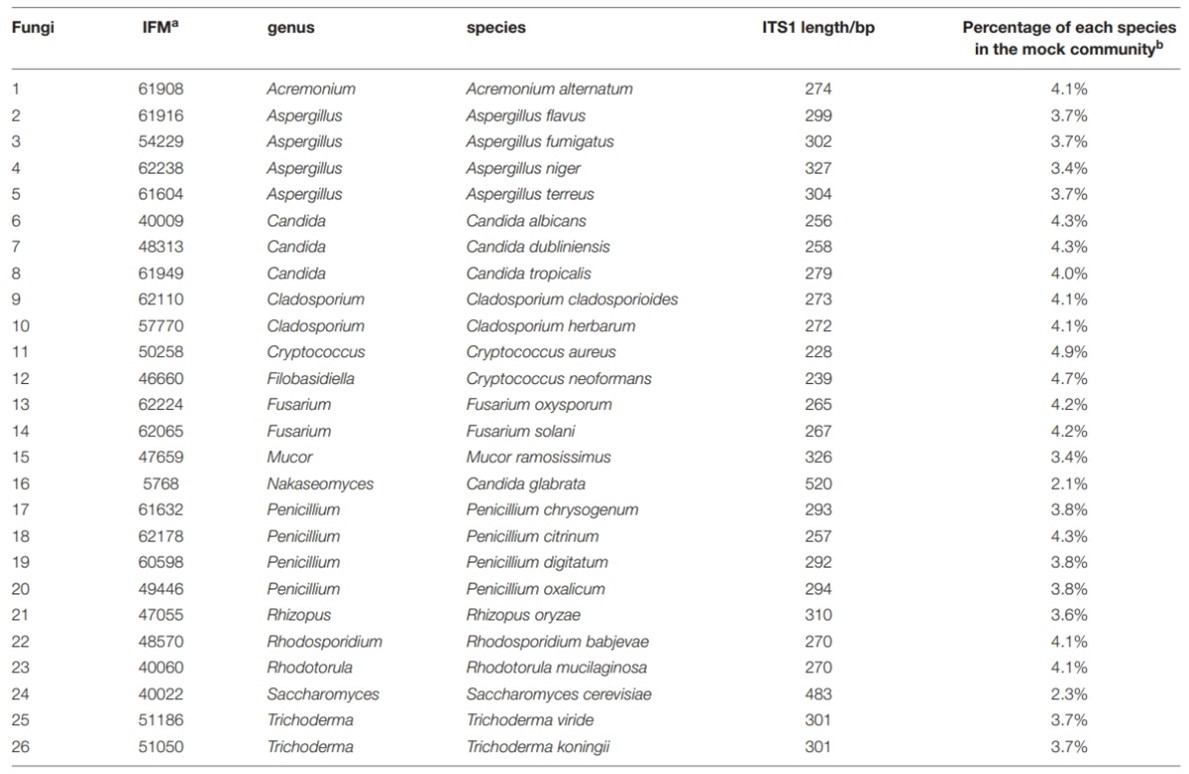
【表一】
以IonPGM數據來看,Nakaseomyces、Saccharomyces這兩個屬的定序結果比起mock真實百分比,不到原先應有量的10%,而Filobasidiella的佔比為mock的兩倍。有四個屬的真菌在不同MQV (mean quality value) 標準時,其在標準品中的百分比會有大幅波動的狀況,在MQV20時佔比增加、隨著靠近MQV30時遞減 (圖1A)。若以每屬菌的ITS1長度去分析百分比時,標示為深紅色、擁有較長ITS1片段的Saccharomyces和Nakaseomyces幾乎在分析中無法被偵測到 (圖1B)。分析讀長與MQV值之間的關係時,在讀長50 bp時為MQV20,而片段長度增加至275 bp時,降至MQV15 (圖1C)。因其真菌菌落百分比會隨著MQV值波動,使得在層次聚類分析上,只有特定幾個MQV標準之下,會與標準品組成較相似 (圖1D)。

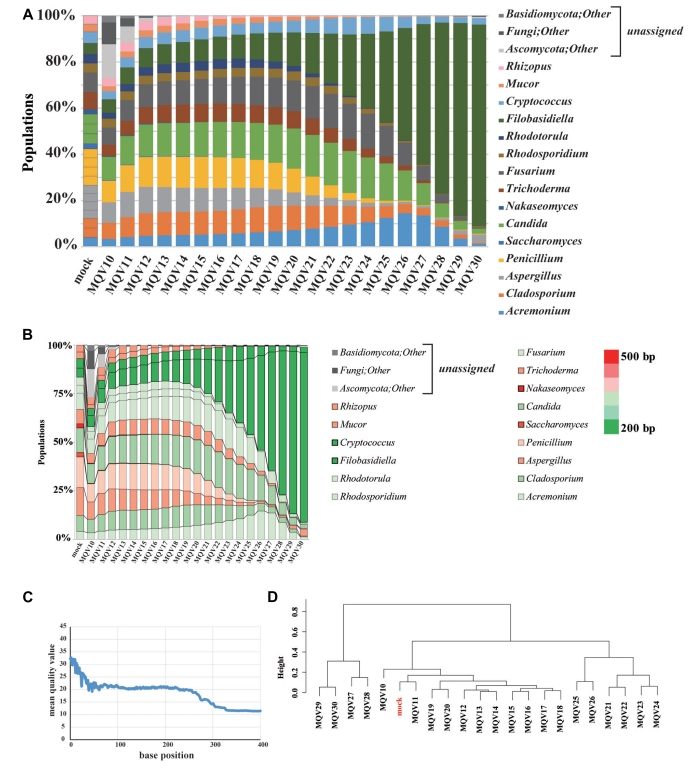
【圖一】
以Miseq數據來看,Nakaseomyces這個屬無法被偵測到,Saccharomyces的比例為mock的一半,其他有三個屬的真菌其百分比偏離原mock的30%左右 (圖2A)。雖不像IonPGM一樣,會因MQV值的不同影響真菌的豐度,但也有因讀長增加MQV值降低的現象發生 (圖2B),但所有的MQV值結果在親源上與標準品是相近的 (圖2C)。
.jpg)
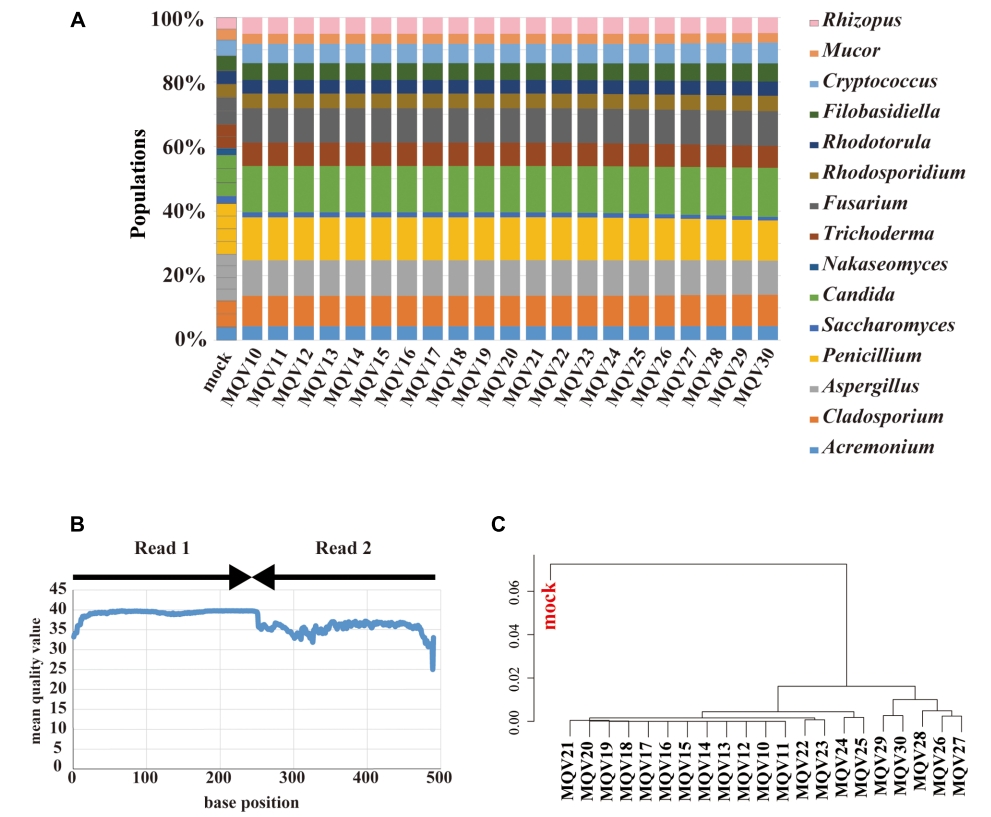
【圖二】
以PacBio數據來看,其分析結果與MiSeq相同,不因MQV值影響各真菌佔比,且所有的屬都可以被偵測到 (圖3A)。使用CCS (circular consensus sequencing mode)模式可以增加其MQV值,當讀長超過300 bp時,CCS的full-pass越多,能提供越高的MQV值。讀長為520 bp時,4個full-pass可達MQV30、8個full-pass可達MQV40,在8個full-pass的條件之下,可以辨識mock中98%以上的真菌 (圖3B)。就層級的聚類上,不管是在多少full-pass的情況下,PacBio皆能夠提供精準的評估 (圖3C)。


【圖三】
可以從這篇文獻中發現,三代PacBio平台的定序結果,分類上比起二代IonPGM或MiSeq更貼近標準品 (圖4C)。總結來說,以真菌標準品定序來說,不同於二代定序,三代定序能完整將標準品的所有物種定序出來,不會特別遺漏ITS1片段較長的物種,且定序出來的量化結果也較像標準品真實原貌 (圖4A-B)。

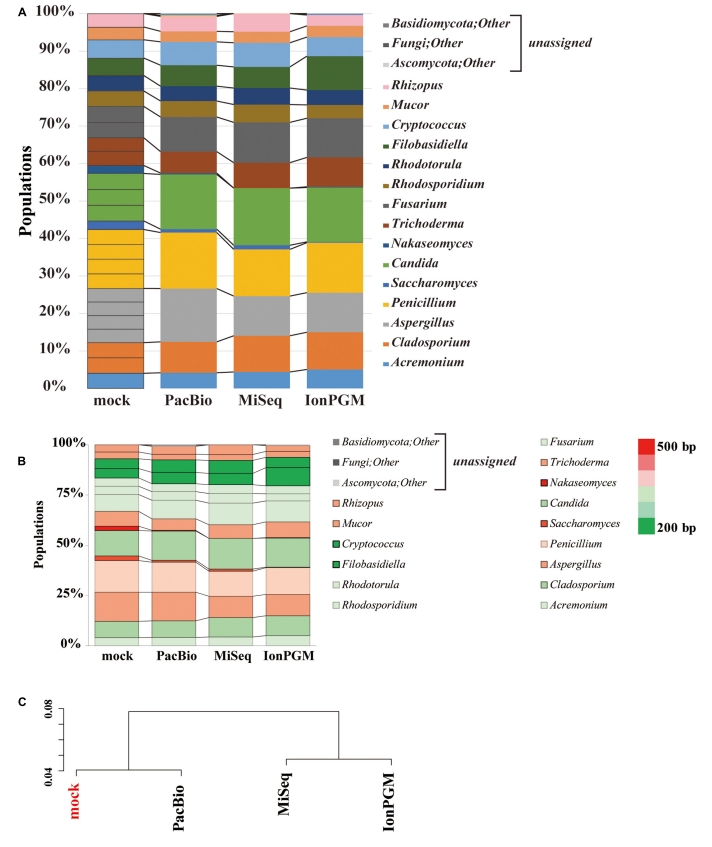
【圖四】
【原文連結】Fungal ITS1 Deep-Sequencing Strategies to Reconstruct the Composition of a 26-Species Community and Evaluation of the Gut Mycobiota of Healthy Japanese Individuals >link
>了解更多三代定序服務
圖爾思生物科技 / 微生物體研究中心
許瑄珉 文案



