03
2019.04
RNA-seq 分析工具大比拼
原創文章 引用請註明出處
RNA-seq 已是非常成熟且廣為使用的技術,各種分析工具也不斷的被發展、進化與發表。2017 年在 Nature Communications 上發表的文章『Gaining comprehensive biological insight into the transcriptome by performing a broad-spectrum RNA-seq analysis』以 short- (illumina) & long-read (PacBio) 對 15 個樣品 (正常人類樣本、乳癌細胞、人胚胎幹細胞) 利用 39 種分析工具進行 120 種分析組合流程,完成 490 次分析,並以 SEQC (Sequencing Quality Control Consortium) 的檢測結果作為對照,基於結果的準確度與分析時間來評估最佳分析方法、工具與組合流程 [1]。另外有三篇分別發表在Breifings in Bioinformatics, PeerJ 與 PLoS ONE 的論文,探討了 normalization 方法的好壞、生物學重複 (Biological Replicate) 及定序深度的重要性及適用工具 (亦可參考先前文章:生物學重複與定序深度在RNAseq研究中何者重要?)。
以下就從每個步驟來探討比較摟~~
序列比對 (HISAT2 / TopHat / STAR)
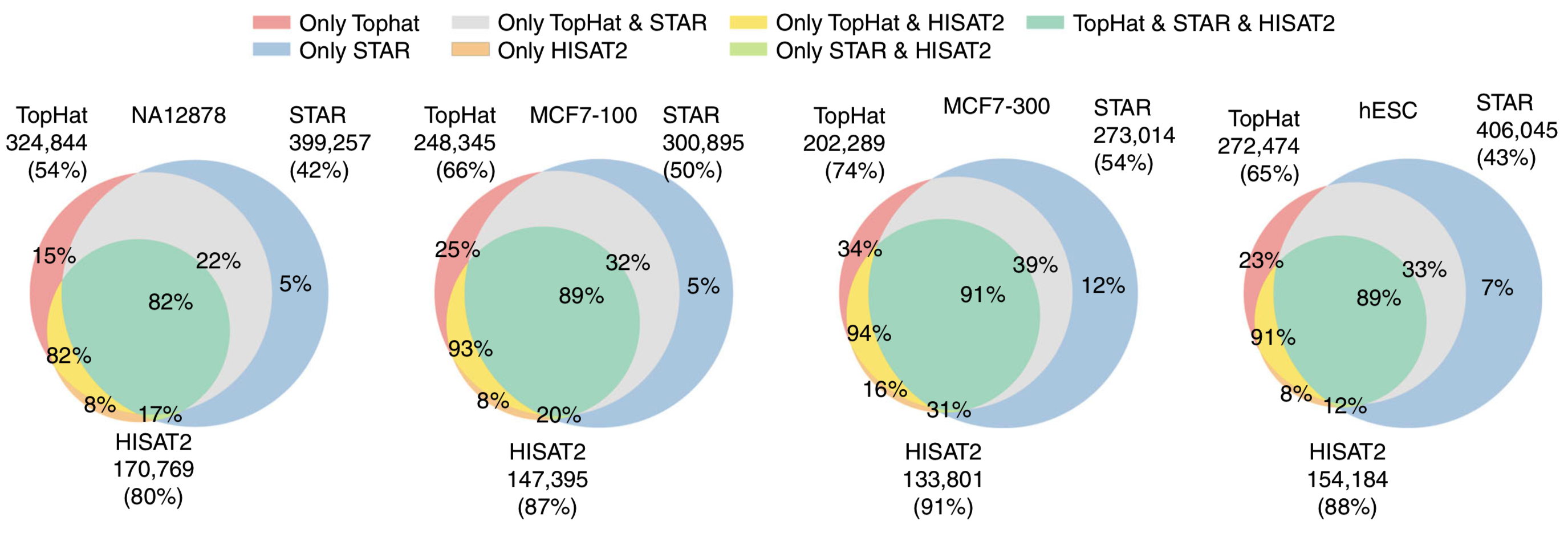
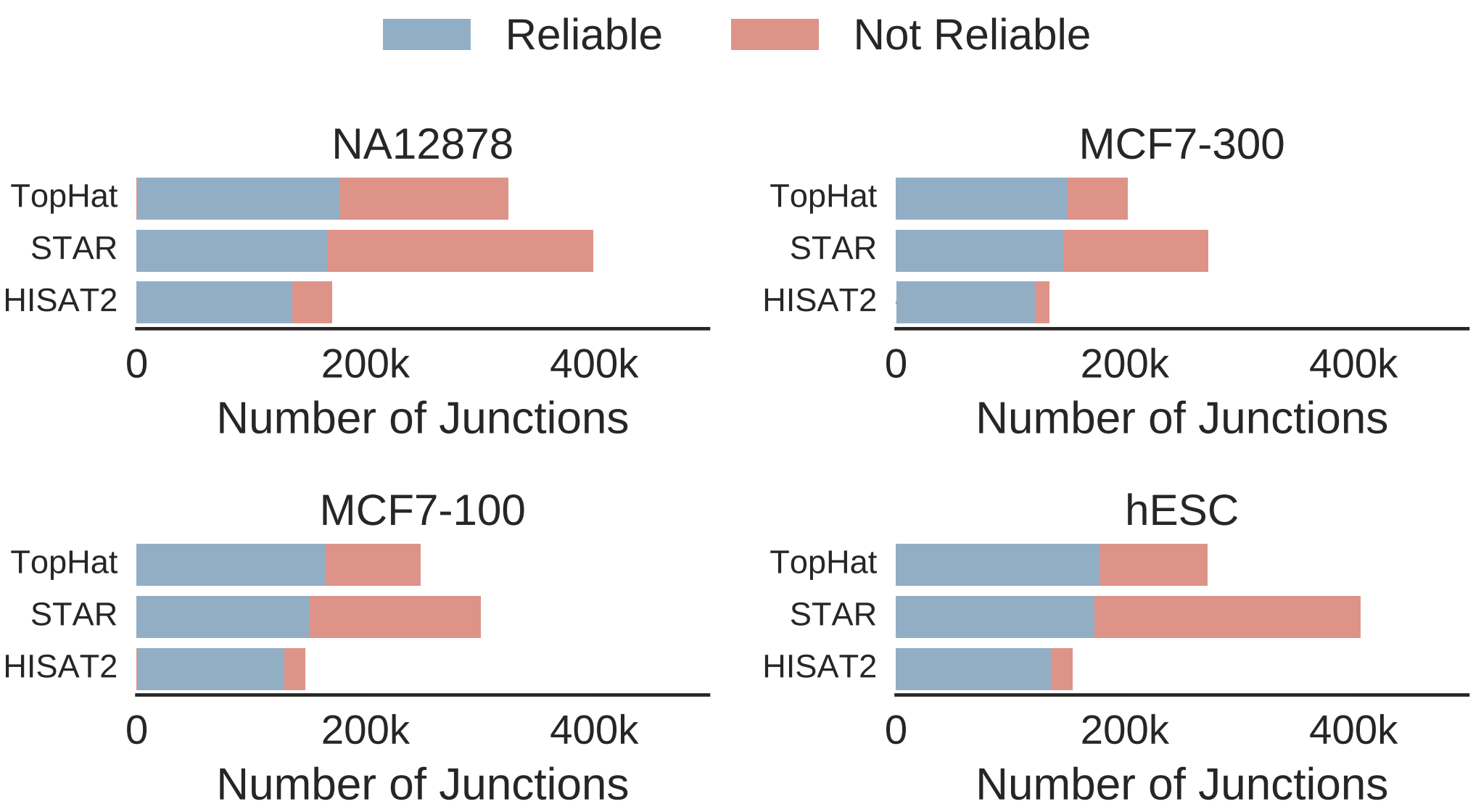
RNAseq reads 比對不同於基因體 reads 比對的地方在於比對的 reads 可能來源於兩個被 intron 隔開的 exon 區域,導致 reads 一端比對在第一個 exon 後面,另一端比對到第二個 exon 的前面,而形成 exon-exon junction。論文中以 Splice junctions 偵測利用 dbEST 資料庫中鑑定到的可靠 junctions 的一致性衡量工具的準確性。Venn Diagram 的圓圈大小表偵測到的 junction 數目,並對每個工具顯示出 junction 數目及驗證率。可以發現 TopHat 和 STAR 雖然在數量上高於 HISAT2,但 HISAT2 在自己獨有的 junctions 是最少的,且 HISAT2 的驗證率皆有 80% 以上 (STAR 在 42%~54%)。
從驗證率來看:HISAT2 > TopHat > STAR
(2) STAR 僅保留雙端比對上的序列,HISAT2 & TopHat 則可進行單端比對。雙端唯一比對比例以 STAR 最高,HISAT2 略為次之。在 soft-clipped 與 mismatches 比例上,STAR 結果最差、HISAT2 最佳。(TopHat 不允許 soft-clipped)
(藍色) 雙端唯一比對 ; (黃色)雙端多重比對 ; (綠色)一端唯一/一端多重比對
(橙色) 一端唯一比對 / 一堆比對不上 ; (紫色) 一端多重比對 / 一堆比對不上 ; (紅色) 雙端皆比對不上
從 soft-clipped 與 mismatches 比例來看:HISAT2 > TopHat > STAR
(3) 比對速度 TopHat 在所有樣品速度皆是最慢,平均看來至少比 HISAT2 慢五十倍以上。(表格單位:小時)
從速度快慢來看: HISAT2 > STAR > TopHat
基於參考基因體的組裝 (Cufflinks / StringTie / Iso-Seq, 三代 / IDP, 二代+三代) 轉錄組裝評估依據 GENCODE v19 參考轉錄體註釋,不在其中的 transcript 視為 false positive。每個 transcript 中包含的 exon 數目是組裝好壞的評判標準,通常單個 exon 的 transcript 可信度最低。Cufflinks 的單exon transcript 約佔 30-40%,StringTie 約 15-20%。StringTie 得到的 transcript 數目約為 Cufflinks 的兩倍,且 exon 數目的分佈比例與 GENCODE v19 較為相似。而 IDP 組裝所得都是多 exon transcript。
從單個 exon transcript 來看:IDP > StringTie > Cufflinks 在 gene level 的組裝,IDP的準確性與靈敏度皆為最佳,而 Cufflinks 則比 StringTie 更為準確和靈敏。與比對工具的組合來看,在 MCF7-300 可發現不管是 Cufflinks 或 StringTie,搭配 STAR 的準確性和靈敏度皆略低於 TopHat 與 HISAT2。在 transcript level,IDP 的準確性比其他工具皆高出20%以上,但靈敏度低於 StringTie、略高於 Cufflinks。而 StringTie 在 transcript level 的精確性和靈敏度皆高出 Cufflinks 15-25%。
從 gene Level 精確性與靈敏度來看:IDP > Cufflinks > StringTie (搭配 HISAT2 / TopHat > STAR) 從 transcript level 精確性與靈敏度來看:IDP > StringTie > Cufflinks (搭配 HISAT2 / TopHat > STAR)
表現定量
通常表現量分析是將 reads 比對參考基因體,若研究想看的是已知和新的transcript,則比對後進行組裝評估表現; 若只想訂量已註釋的基因,則直接比對後在使用工具做 normalization 進行評估 (基於基因體比對:cufflinks & StringTie; 基於 transcript 比對:eXpress & Salmon-Aln)。論文中還比較了不需比對直接判斷 (alignment-free) read 來源於哪個 transcript 的方式 (Sailfish、Salmon、quasi-mapping & kallisto) 以及基於 long-read 技術的 IDP。
根據 Spearman Correlation 熱圖分析,Cufflinks 的定量結果與其他工具相關性最差 (低於0.4),不需比對直接定量的工具與 StringTie 結果更接近。整體看來,StringTie 相對於 Cufflinks 一致性較好。
對於同一個樣品不同 read length (MCF7-100 & MCF7-300) 的分析比較可以看出比對工具定量的穩定性。由圖中可見 (越集中於 log2 FC = 0 定量一致性越好),IDP 具有高度一致性,而 short read length 中,HISAT2 的一致性最好。其中還可發現,若採用 STAR 搭配 Cufflinks 與 StringTie,其表現量結果不穩定 (左二 & 左五)。
從樣本特異性與read長度偏好性來看:HISAT2 > TopHat > STAR
Normalization 歸一化方法 (TC / UQ / Med / DESeq / TMM / Q / RPKM / RawCount) 目前研究上已漸漸不採用 FPKM/RPKM 來做 normalizatoin,那究竟該選用什麼方式最穩定/最可靠呢?我們來看看發表在 Brief Bioinformatics 的一篇比較性文章的評估 ~比較中使用八種方式對小鼠七種條件和重複數據做 normalization 分析,除了 TC 、RPKM 與 RawCount 穩定度不佳其餘方法都較為穩定且相似。對10組模擬資料的 False-positive rate 可見,DESeq 與 TMM 的表現最好且穩定 [2]。(DESeq/DESeq2 normalization 稱為 Relative log expression, RLE)
因此綜合比較下,採用 DESeq 與 TMM 做 normalization 最穩定可靠。
差異表現 (DESeq2 / edgeR / limma / sleuth / CuffDiff / Ballgown)
RNA-seq 的首要目的就是找出不同分組樣品間的差異表現基因,比較中藉由 SEQC 樣品 (SEQC-A vs SEQC-B, SEQC-C vs SEQC-D)中1001個有 qRT-PCR 定量的基因作為對照評價。整體平均來看,DESeq2 在所有組合中表現最好,sleuth、edgeR 和 limma 略差,CuffDiff 和 Ballgown 準確度遠低於基於 read count 工具的準確度。AUC-30 的分析,edgeR 與 DESeq2 表現最佳。另外,從速度上來看的話 CuffDiff 是最慢的工具。
從 qRT-PCR 對照評價來看:DESeq2 > edgeR > limma >> sleuth > CuffDiff > Ballgown
生物學重複穩定性
另一篇發表在 PeerJ的論文,也比較了多種方法 (DESeq2 / DESeq / edgeR / voom / Z-test / NOISeq / GFOLD),結果顯示 DESeq2 隨著生物學重複數增加其 PPV (positive predictive value) 與靈敏度取得了最佳平衡 (n=3時,平均 PPV 52.5 ± 10.8% 平均靈敏度 36.0 ± 5.7%; n=6時,平均 PPV 62.1 ± 7.7%,平均靈敏度 65.1 ± 4.5%),另外 edgeR 也表現合理的結果 [3]。
在 PLoS ONE 的一篇文章進行了三種工具於生物學重複數量的比較 (DESeq / CuffDiff2 / edgeR) [4],當重複數量從 2 增加至 20 時,由 ROC curve 可知 edgeR 略優於 DESeq,CuffDiff2 則表現最差。
不同定序深度穩定性
從定序深度的不同比較發現,CuffDiff 對於定序深度的不同最為敏感,而 DESeq 與 edgeR 在不同深度時保持穩定的表現 (DESeq 穩定度略優於 edgeR)。因此當定序深度不深時,建議採用 DESeq & edgeR。
從定序深度的不同比較發現,CuffDiff 對於定序深度的不同最為敏感,而 DESeq 與 edgeR 在不同深度時保持穩定的表現 (DESeq 穩定度略優於 edgeR)。因此當定序深度不深時,建議採用 DESeq & edgeR。
從生物學重複的適用性來看:DESeq2 > edgeR > Others 從定序深度來看:DESeq2 > edgeR > Others 這次小編策劃的專題報導目的是希望能以大規模比較的方式,討論出一個在RNA-seq分析 (基於參考基因體) 時的最佳流程方案,畢竟做研究還是要採用最好且被信賴的工具,未來,有更新的工具出現小編會繼續努力跟上並分享給大家知道!!
參考文獻
[1] Sahraeian, S. M. E., Mohiyuddin, M., Sebra, R., Tilgner, H., Afshar, P. T., Au, K. F., et al. (2017). Gaining comprehensive biological insight into the transcriptome by performing a broad-spectrum RNA-seq analysis. Nature Communications, 8(1), 1–14.
[2] Dillies, M.-A., Rau, A., Aubert, J., Hennequet-Antier, C., Jeanmougin, M., Servant, N., et al. (2013). A comprehensive evaluation of normalization methods for Illumina high-throughput RNA sequencing data analysis. Briefings in Bioinformatics, 14(6), 671–683.
[3] Khang, T. F., & Lau, C. Y. (2015). Getting the most out of RNA-seq data analysis. PeerJ, 3(Suppl 9), e1360–20.
[4] Zhang, Z. H., Jhaveri, D. J., Marshall, V. M., Bauer, D. C., Edson, J., Narayanan, R. K., et al. (2014). A Comparative Study of Techniques for Differential Expression Analysis on RNA-Seq Data. PLoS ONE, 9(8), e103207–11.
圖爾思生物科技 / 微生物體研究中心
郭育倫 文案