22
2021.10
【RNAseq 3小學堂】差異基因表現量分析_任你客製的Heatmap
原創文章 引用請註明出處
#RNAseq #差異基因 #雲平台 #3小學堂
|| 3分鐘速解RNAseq分析,本周介紹超高自由度Heatmap ||
觀察基因表現量差異除了上篇 提到的MA plot和Volcano plot之外,大家最熟知的另一個圖就是 #Heatmap !
Heatmap 可以更直觀的觀察基因表現差異,但當拿到分析圖時,總會想,如果我....調整會不會更好呢? 來來回回跟廠商溝通改圖超麻煩,真想自己來,但不會寫code怎麼辦?!
|| 圖爾思雲平台,讓你輕鬆調整 ||
小編將所有客製化功能分成4大類:初始設定、聚類、顏色、大小
A. 初始設定
想先得到第一張Heatmap前,有6點你需要先進行設定
-
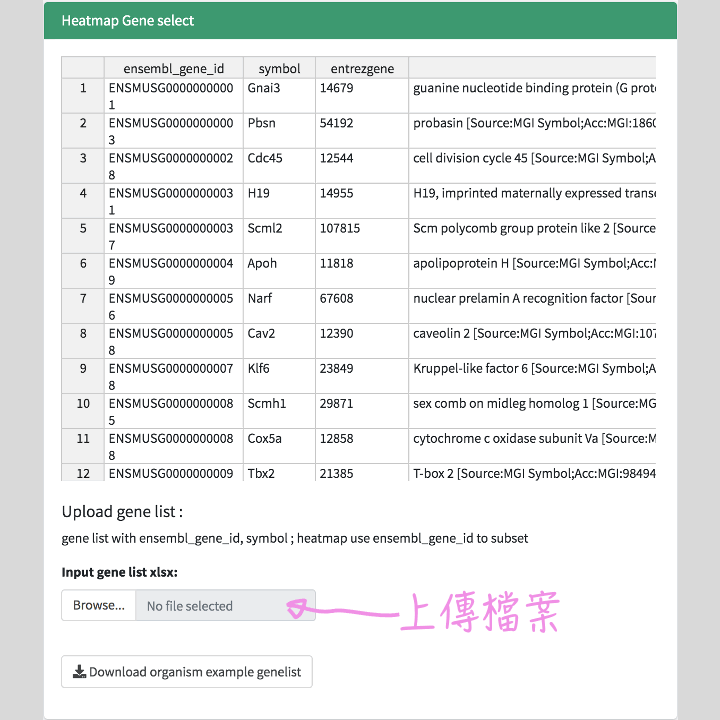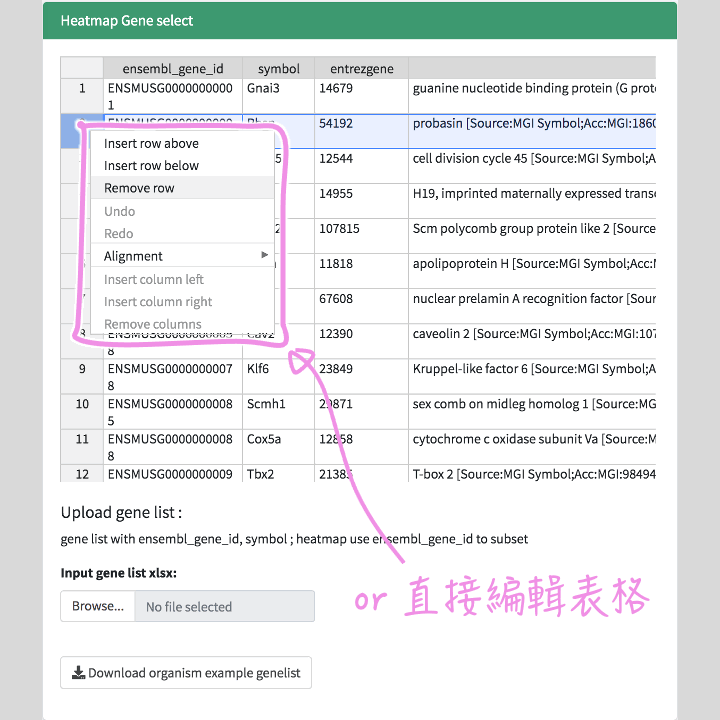
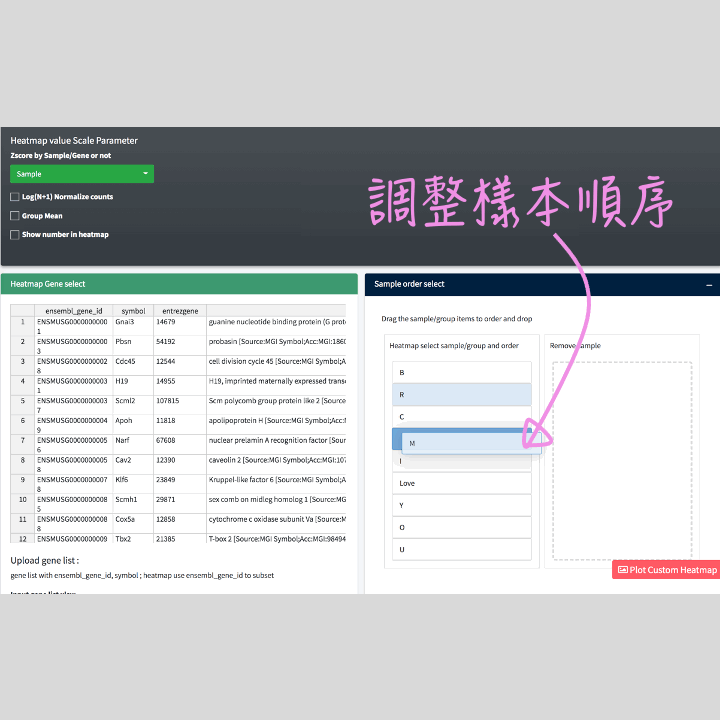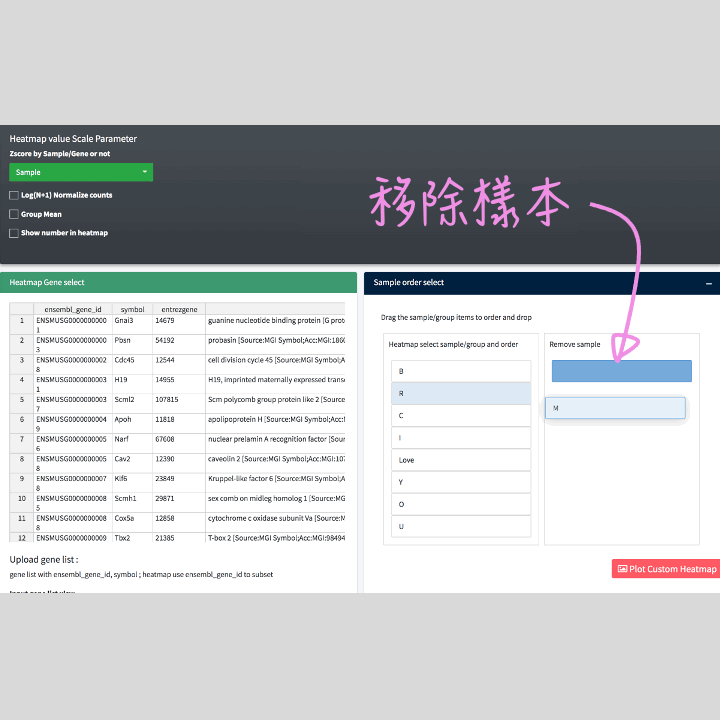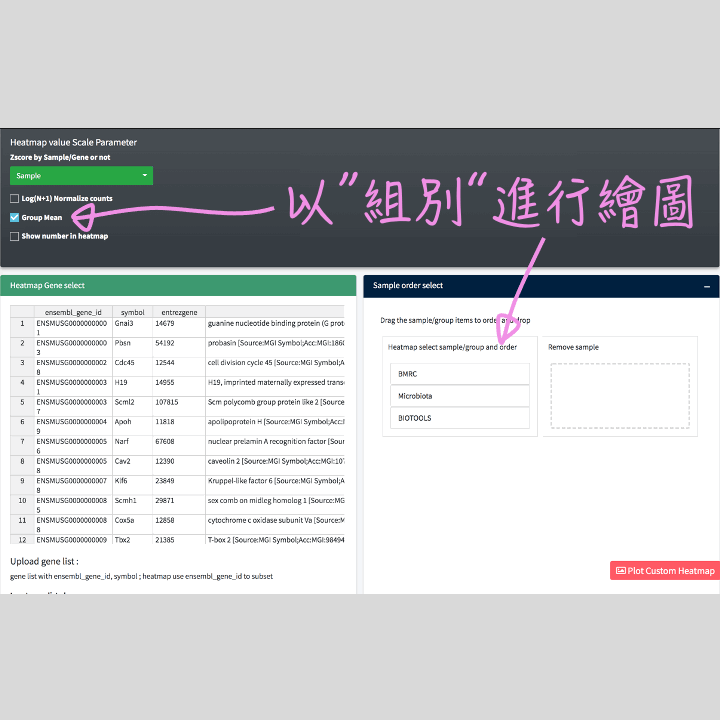
基因列表:初始進入到客製化介面時,就會先提供您再DEG Analysis所設定的篩選結果 。若想再調整列表,可以在下圖標示的位置上傳你的基因列表,或是直接編輯表格,基因列表需要包含 ensembl_gene_id, symbol 兩個欄位資訊,也可以直接上傳前一個階段分析下載的 DEG 結果列表。
-
樣本與組別:可以選擇以Group mean 進行繪製。想要快速篩選要與不要的組別/樣本,你可以在”Sample order select”,自由 保留/移除 和 移動組別/樣本 的順序。
-
標準化方法:提供3種標準化方法任你選 TPM、FPKM、DESeq2 (RLE) (有生物重複)/edgeR (TMM) (無生物重複),也可以選擇標準化後的數值是否取Log。取Log的好處於,可避免heatmap在顏色的呈現上受到極端值的影響。
-
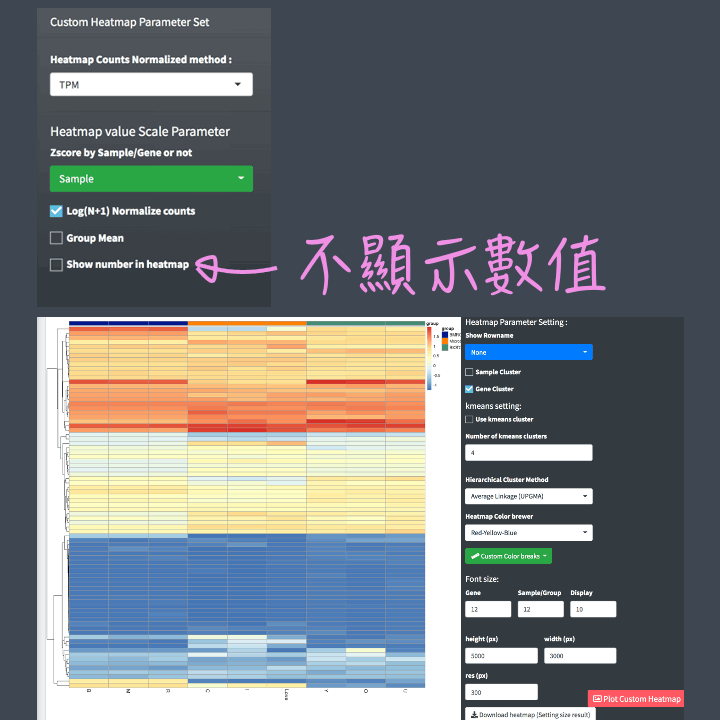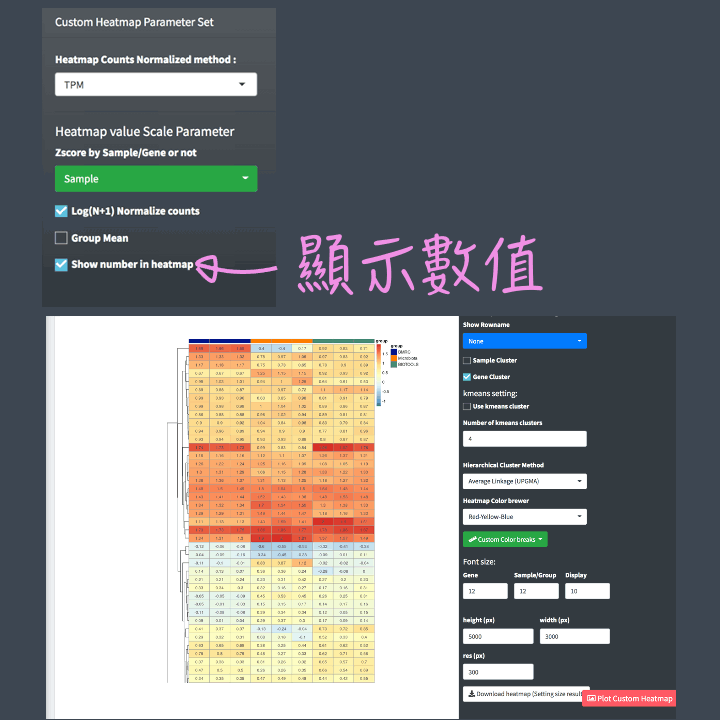
數值呈現:可以選擇是否在Heatmap上秀出heatmap value。若不想顯示heatmap value在熱圖上,但仍想要確認熱圖各個位置的數值,也可在Heatmap Value Table 確認與下載對應數值喔。
-
Zscore:zscore的設定可以避免極端值影響圖上顏色的呈現,將變數轉為常態分佈。可以選擇zscore的方向(基因 或 樣本),方向不同,對結果的解釋方式就不同。
-
基因呈現:繪製熱圖時,可以是否選擇顯示基因名稱,可以選擇顯示ensembl_gene_id 或是 symbol 名稱資訊。
B. 聚類設定 (Clustering)
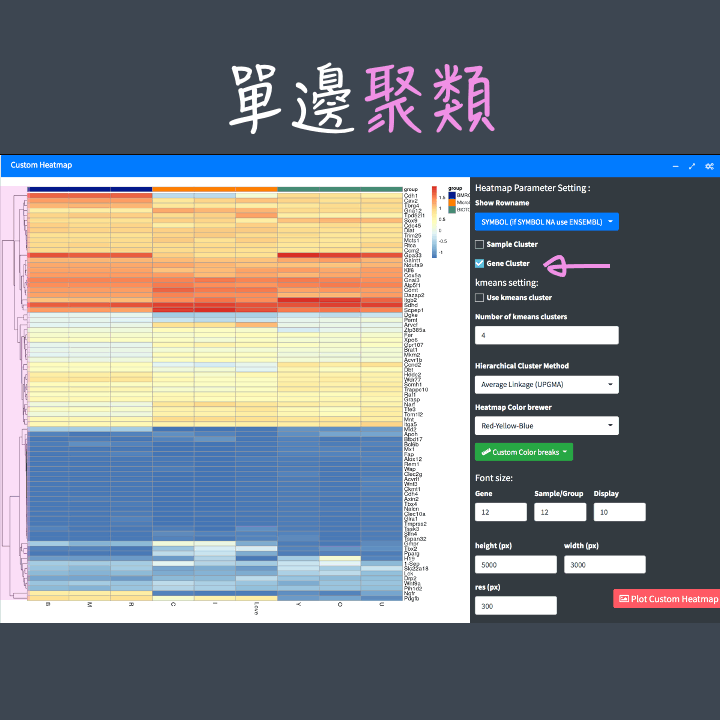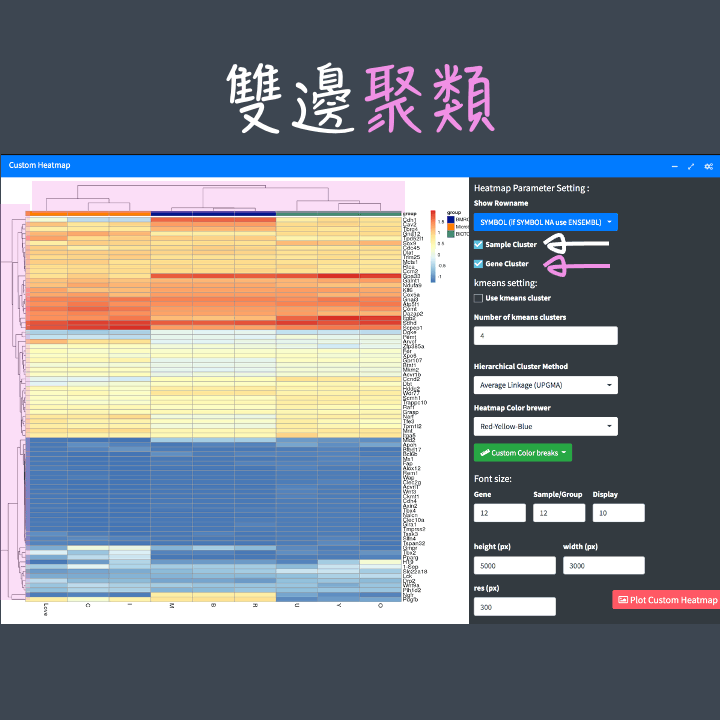
聚類的目的是要將表現量相近的 樣本/基因 聚類在一起,以利更直觀的觀察結果。
-
方向:可以選擇單邊聚類 (樣本 或 基因) 或 雙邊聚類 (樣本 和 基因)。
-
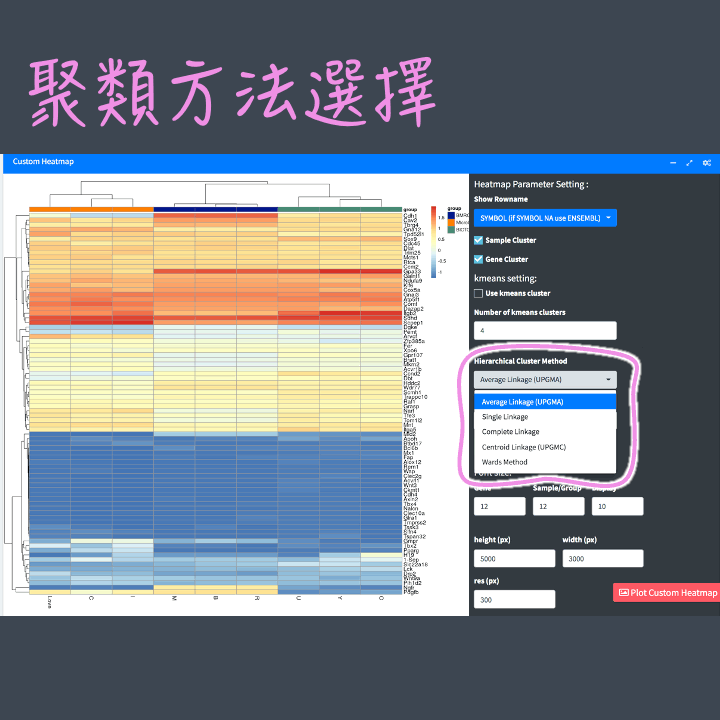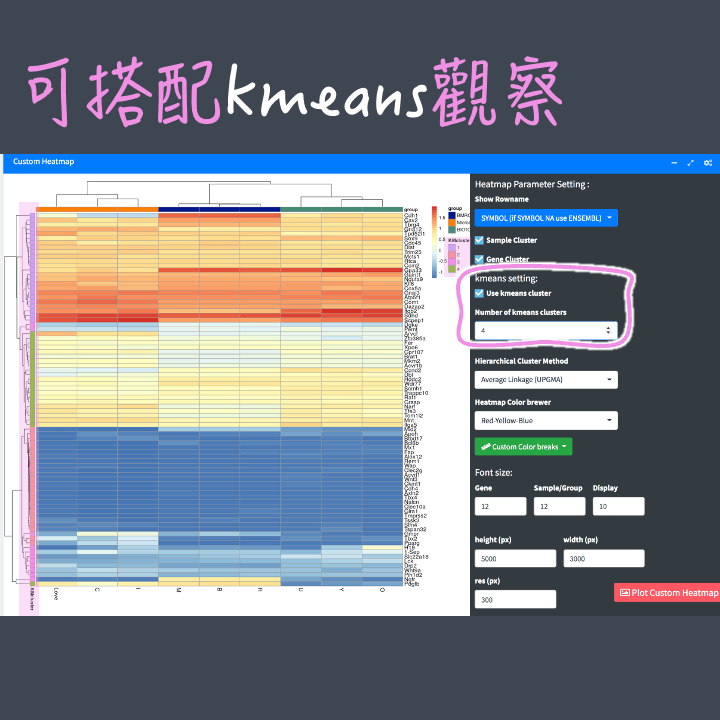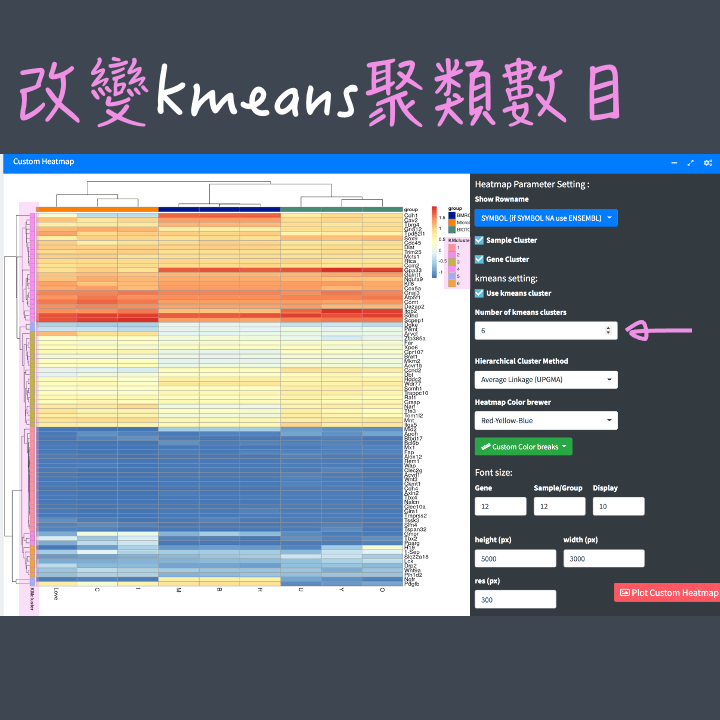
聚類方法:有共6種聚類方法可以選擇,其中 kmeans cluster 須再設定"數量",因此設定的位置和另外5種的位置不同喔! kmeans cluster 會依照設定的群數給予分群的基因顏色,也可同時搭配其他5種聚類樹觀察結果。
C. 顏色
-
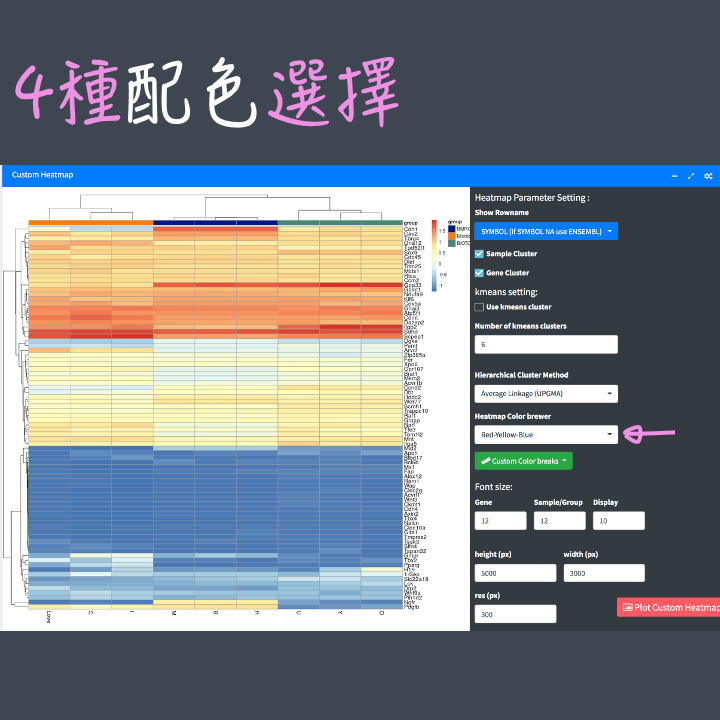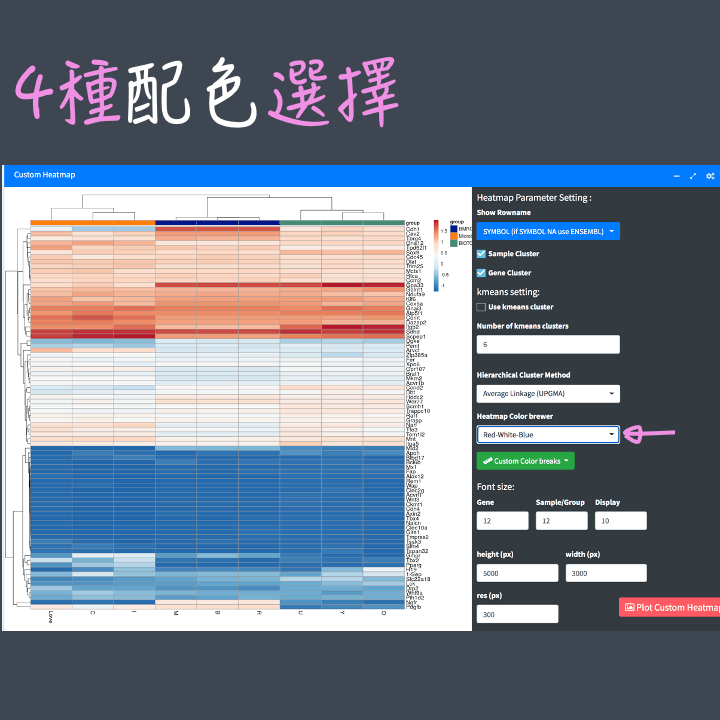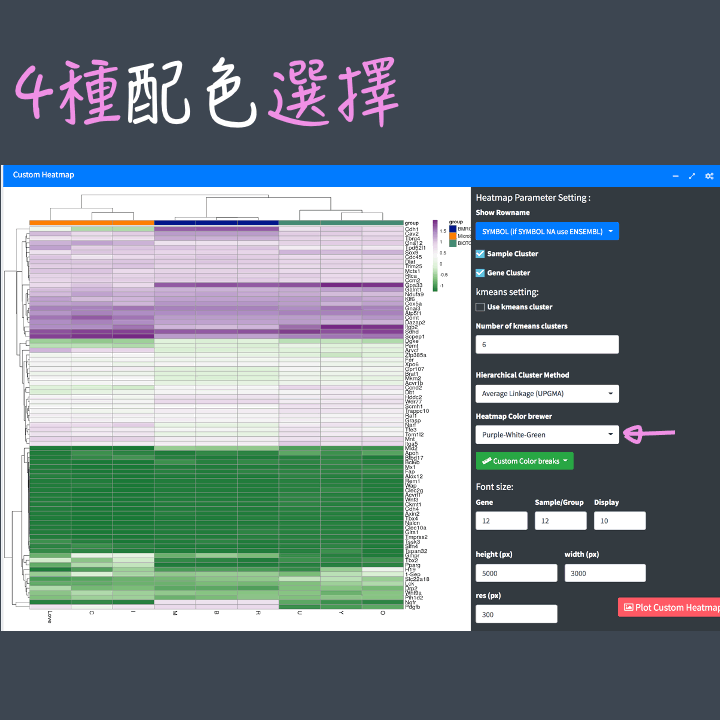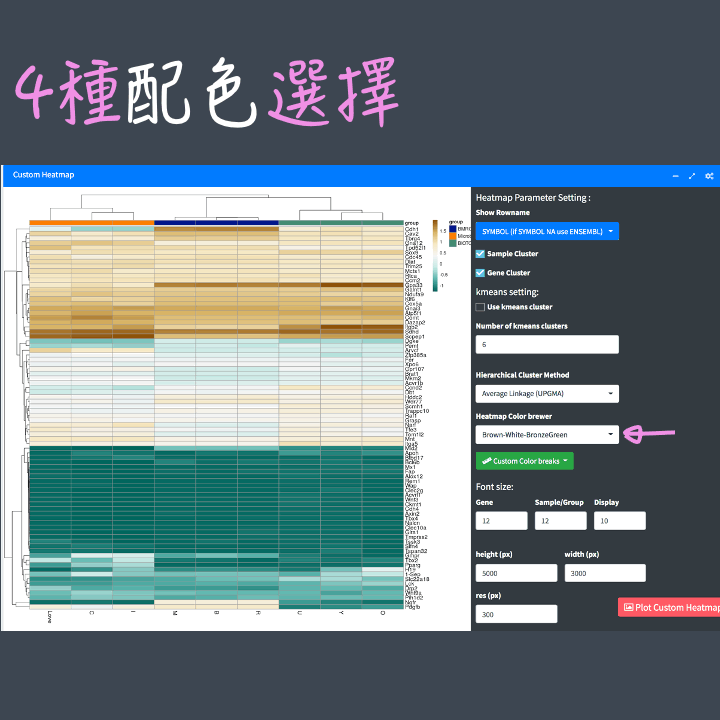
Heatmap配色:不愛紅-黃-藍配色沒關係,雲平台提供你4種配色自由選。
-
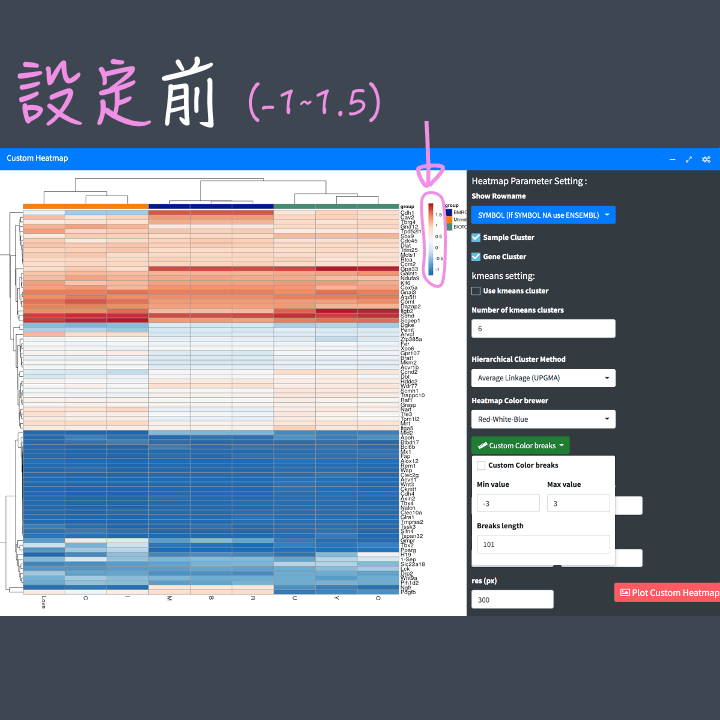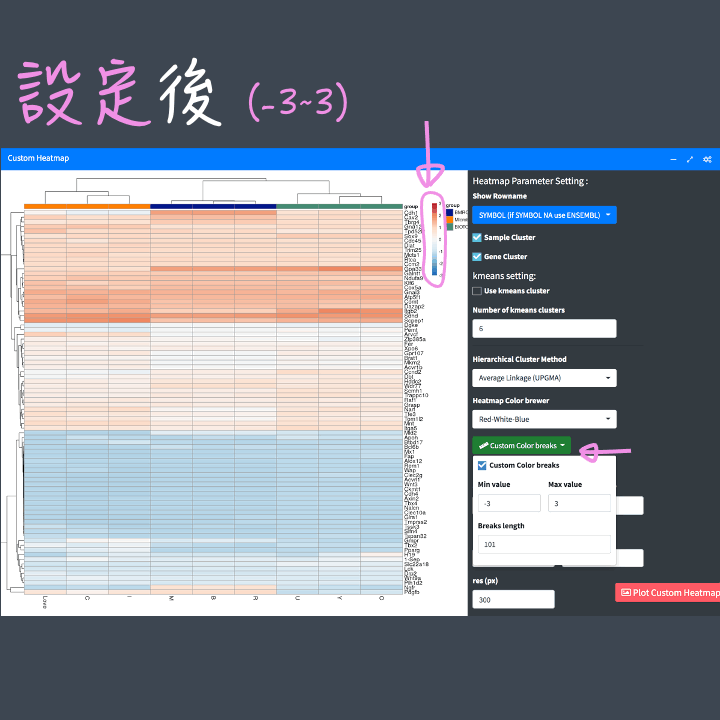
Color breaks:可設定Heatmap value的最高最低值,該值的設定會影響顏色的表現,可見下方圖的示範。
-
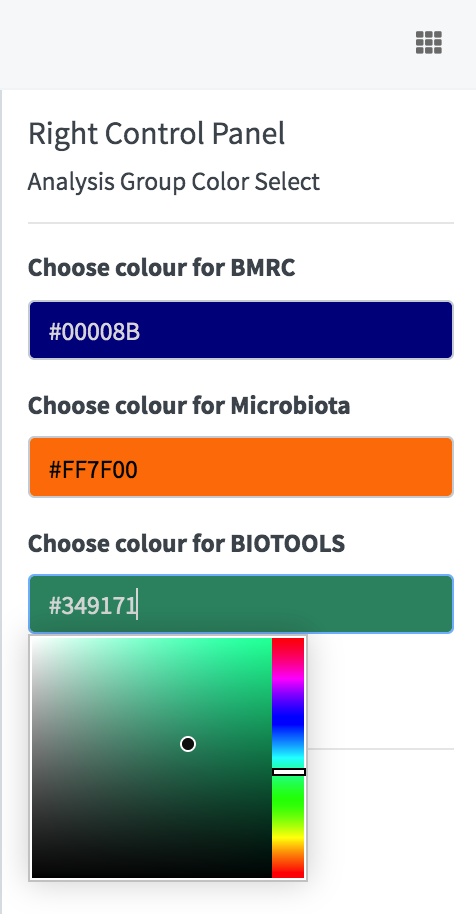
組別顏色:為你的組別自由配色。
D.大小
-
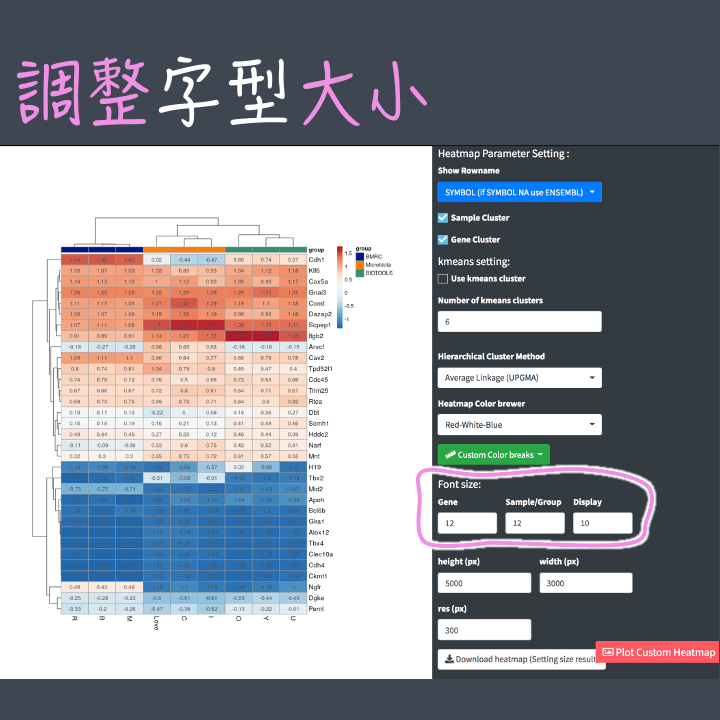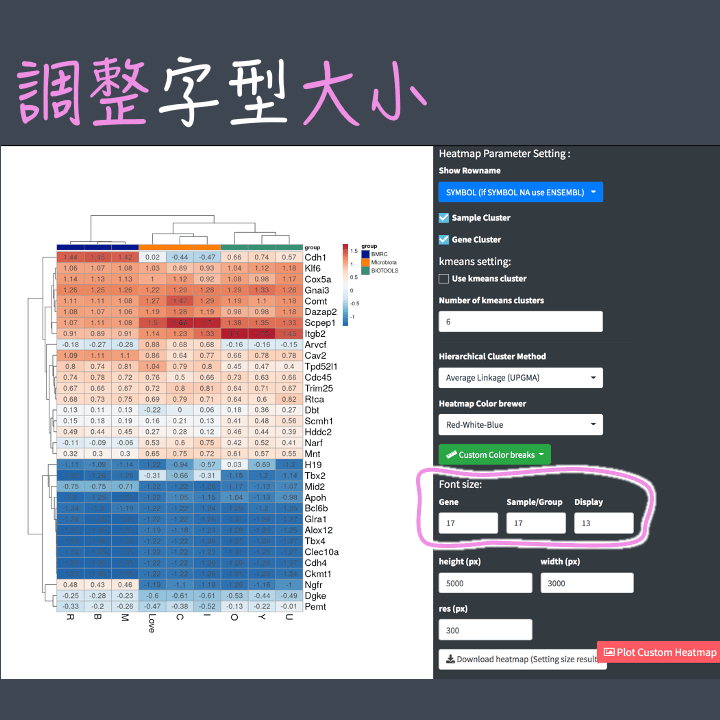
XY軸字體:調整 基因、樣本、每格heatmap value 的字體大小。
-
圖片:自由調整圖片長、寬、解析度,調整後還能下載保存喔!
很多人會問小編:
|| 有這麼多標準化、聚類等計算方法可以選,我到底要怎麼選? ||
在雲平台上的計算方法,都是文獻中有被使用的統計方法,多方嘗試與篩選,找出利於你解說實驗故事的統計方法,就是適合你的計算方法。
雲平台讓你在任何時間、任合地點,只要有電腦、有網路、有瀏覽器,想怎麼調、就怎麼調 !
圖爾思生物科技/微生物體研究中心
沈筱凌/顏維萱
沈筱凌/顏維萱