14
2016.09
三代定序在微生物研究的應用
這次就跟大家聊一聊新技術在微生物研究中的應用。細細想來,新技術的應用,怎麼能少得了它—全長擴增子定序。
定序平台
先介紹一下定序平台,目前應用較廣的技術是 PacbioRS II 和 Nanopore,Nanopore 利用新型納米孔技術,依據檢測DNA序列通過納米孔時電流的變化判斷鹼基資訊,但由於納米材料穩定性、電流幅度較小及DNA移位速率過快等問題,目前其應用的接受度沒有Pacbio RS II SMRT 定序高。
擴增子是什麼?
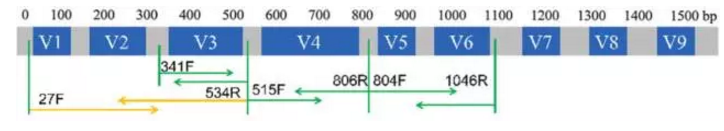
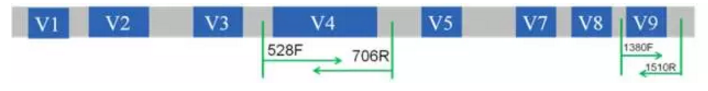
擴增子(amplicon)是指DNA或RNA擴增後得到的一段DNA序列。擴增子定序目前主要用於微生物物種的鑑定,當然應用到高等動植物某些擴增產物的定序上也未嘗不可。主流的擴增子定序技術有16S/18S/ITS三種,其中,16S/18S rRNA的編碼基因rDNA(編碼核醣體RNA小亞基的基因),其結構既具有保守性又具有高度的變異性,高變區被分為9個區域,V1-V9(見圖1)。保守性能夠反映出生物物種間進化的親緣性,高變性可用於物種鑑定,隨著高通量定序技術不斷發展,成本不斷下降,透過對9個高變區的某1~2個區域進行定序來進行物種鑑定,已經成為主流的研究方法。
但由於微生物突變位點的分佈並不均勻,1~2兩個區域的序列資訊並不能完全真實的還原微生物群落情況,而三代定序恰好可以解決這個問題。
但由於微生物突變位點的分佈並不均勻,1~2兩個區域的序列資訊並不能完全真實的還原微生物群落情況,而三代定序恰好可以解決這個問題。
圖1. 16S/18S rDNA 結構示意圖
全長擴增子
那麼全長擴增子定序是怎麼做的呢?上圖簡單明了說明問題,要注意的是,一般情況下,全長擴增利用通用引物實現,但由於擴增子需要資料量較小,為了節約成本都會採用混樣建庫,為了區分不同樣本,在全長擴增子時會加上 barcode(相當於二代定序中加的index)。
圖2. 全長擴增子定序試驗流程圖
數據量
接著~問題來了,全長擴增子定序需要多少數據量比較合適呢?小編查了目前所有的全長擴增子文獻,總結建議如下:簡單樣本定序最低數據量3K條;複雜樣本最低建議5K條,關注低豐度物種數據量不限,建議1W條或更多(當然依照經費預算而定)。
定序錯誤率
再來談談大家關注的另一個問題,三代定序的隨機錯誤率約為20%,究竟準確性如何?小編簡單算了一下,全長16S建完庫約為1.6K,定序時間和DNA聚合酶讀長約為10K,一個read可重複測一個序列6次,CCS校正正確率可以達到99.65 %,當然如果可以達到20X的話,正確率就能達到99.999%,所以大家完全可以不用擔心錯誤率的問題。
優勢及劣勢
做了全長擴增子定序後可以得到哪些資訊呢?更長的讀長更完整的擴增區域可獲得:一,更準確的物種分類;二,更多更準確的物種註釋信息。當然,缺點也很明顯:較貴,大約需要耗費二代定序約10~15倍的價格,當然這也跟定序數據量的大小有關。
參考文獻
[1] Singer E, Bushnell B, Coleman-Derr D, et al . High-resolution phylogenetic microbial community profiling[J]. IsmeJournal , 2016.
[2] J Shin,S Lee,MJ Go,YL Sang,CK Sun,et al . Analysis of the mouse gut microbiome using full-length 16S rRNA amplicon sequencing[J]. Scientific Rports , 2016.
[3] Erin B Fichot, R Sean Norman. Microbial phylogenetic profiling with the Pacific Biosciences sequencing platform[J]. Microbiome , 2013.
[3] Erin B Fichot, R Sean Norman. Microbial phylogenetic profiling with the Pacific Biosciences sequencing platform[J]. Microbiome , 2013.
圖爾思生物科技 / 諾禾致源文案